Role of Two Methanol Dehydrogenases in Methanol Metabolism of Methylobacterium
-
摘要: 本研究以食葡糖食甲基杆菌MP688(Methylobacterium glucophilum MP688)的吡咯喹啉醌(pyrroloquinoline quinone,PQQ)高产突变株J1-1为研究对象,通过生物信息学分析从基因组上找到两个甲醇脱氢酶(methanol dehydrogenase,MDH)基因mpq0771和mpq2496。考察了Ca2+及La3+对菌体生长、PQQ合成、MDH表达及酶活力的影响,并研究两种酶在甲醇代谢中的作用。结果显示:Ca2+和La3+均能促进J1-1菌体生长,且在菌体快速生长的情况下导致PQQ合成下降;Ca2+存在时,菌株J1-1中mpq0771表达量较高,添加La3+后mpq0771表达受到阻遏,但增强了mpq2496表达,菌体MDH总活力也有所提高;敲除菌J1-1Δmpq0771不能利用甲醇生长,添加La3+后可利用甲醇生长并能合成PQQ,且能检测到MDH活性。mpq0771编码产物是Ca2+依赖的MDH,mpq2496编码产物是严格依赖La3+的MDH,且La3+能够诱导mpq2496表达并替代mpq0771发挥甲醇脱氢酶的作用。Abstract: In this study, the pyrroloquinoline quinone (PQQ) high-yield mutant J1-1 of Methylobacterium glucophilum MP688 was used to do bioinformatics analysis to extract the two methanol dehydrogenase (MDH) genes from the genome, mpq0771 and mpq2496. The effects of Ca2+ and La3+ on bacterial growth, PQQ synthesis, MDH expression, and enzyme activity were explored, as well as the roles of the two enzymes in methanol metabolism. The results showed that Ca2+ and La3+ both increased J1-1 bacteriophage growth and decreased PQQ synthesis in the context of fast viral development. In the presence of Ca2+, mpq0771 expression increased in strain J1-1. La3+ inhibited mpq0771 expression while promoting mpq2496 expression and increasing total bacterial MDH viability. The knockout bacteria J1-1Δmpq0771 could not grow in methanol, but with the addition of La3+, it could grow in methanol and synthesize PQQ, and MDH activity was identified. The product encoded by mpq0771 was a Ca2+-dependent MDH, and the product encoded by mpq2496 was a La3+-dependent MDH, and La3+ could increase the production of mpq2496 and replace mpq0771 as a methanol dehydrogenase.
-
Keywords:
- Methylobacterium /
- methanol dehydrogenase /
- pyrroloquinoline quinone /
- lanthanide
-
吡咯喹啉醌(pyrroloquinoline quinone,PQQ)是一种不同于烟酰胺和核黄素的第三类氧化还原酶的辅酶[1],具有增强抗氧化酶活性、促进生成神经生长因子、抗辐射、预防慢性心力衰竭、抗高原缺氧等作用[2−5],在功能性食品和医药行业都具有良好的开发前景。目前,合成PQQ的方式主要有化学合成法和微生物发酵法[6]。与化学合成法相比,微生物发酵法具有污染小、成本低、产率高等优点,因此在工业化生产中被广泛应用[7]。迄今自然界中只有某些革兰氏阴性菌能够产生PQQ,其中甲基营养型细菌的PQQ合成水平最高[8−9]。甲醇脱氢酶(methanol dehydrogenase,MDH)是甲基营养菌代谢甲醇的关键酶,首先催化甲醇还原PQQ生成PQQH2,并以细胞色素c作为电子受体,将两个电子转移至细胞色素c,在该过程中自由基半醌会将PQQH2再氧化回PQQ[10−11]。
甲醇脱氢酶存在于细胞周质中,有MxaFI和XoxF两种类型[12−13]。MxaFI是由MxaF和MxaI所编码的两个α大亚基和两个β小亚基组成的四聚体(α2β2),其中MxaF的活性位点需要Ca2+和PQQ作为辅基[14−15];XoxF型MDH需要在活性位点上结合镧系元素(如镧或铈等)和PQQ才具有生物活性,实现对甲醇的氧化[16]。HIBI等[17]从含有La3+培养基中生长的细胞中纯化的MDH对应于XoxF,而从Ca2+生长的菌体中纯化得到的MDH与MxaF相对应。在甲醇代谢过程中,MxaFI和XoxF两种类型的MDH存在着某种联系和分工。CHISTOSERDOVA等[16]研究发现,MxaF缺陷菌株不能在甲醇上生长,且认为在甲基营养菌中Ca2+依赖型的MxaF型MDH是甲醇代谢中唯一活跃的MDH;后又发现缺失XoxF1和XoxF2的菌株不能利用甲醇作为碳源和能源生长,且几乎完全丧失甲醇脱氢酶活性,与MxaF敲除菌株的表型相同。随着对甲醇脱氢酶的深入研究,发现La3+能够对甲醇脱氢酶进行调控。NAKAGAWA等[18]向MxaF缺失株的培养基中补充La3+,发现能够恢复菌体的生长,实验表明XoxF在菌株AM1中作为La3+依赖型的MDH参与甲醇代谢。
甲醇脱氢酶对菌株生长及PQQ合成有较大的影响[19],李淼鑫等[20]发现提高MDH的表达水平能够影响PQQ的生物合成水平。孙晓宇等[21]通过突变甲醇脱氢酶的启动子来降低其活性,达到了提高PQQ产量的目的。李大攀等[22]将MDH基因敲除后,MDH的活力、PQQ合成水平及氧化甲醇的能力均下降。本实验室从土壤中分离筛选得到一株PQQ产生菌食葡糖食甲基杆菌MP688,能够以甲醇为唯一碳源进行生长,后经多轮物理和化学诱变得到了高产菌株J1-1。在前期研究结果中,将菌株J1-1中的mpq0771基因敲除后,敲除菌不能利用甲醇生长,与Ca2+依赖的MDH特征相似[21]。现经过基因比对分析,在基因组上找到了另外一个甲醇脱氢酶基因mpq2496,推测可能为La3+依赖的MDH。通过考察菌株生长状态、PQQ合成、MDH表达及酶活等因素,初步分析了mpq0771和mpq2496基因在菌株生长代谢中的作用和关系,为阐明甲醇脱氢酶在食甲基杆菌的生长和代谢产物合成过程中的调控机制提了供理论依据。
1. 材料与方法
1.1 材料与仪器
食葡糖食甲基杆菌(Methylobacterium glucophilum)J1-1、大肠杆菌(Escherichia coli)pTIG、敲除菌J1-1Δmpq0771 本实验室保存;细菌基因组DNA提取试剂盒 北京天根生化科技有限公司;快速质粒小提试剂盒 南京诺唯赞生物科技有限公司;限制性内切酶(10 U/μL)、琼脂糖凝胶DNA回收纯化试剂盒 赛默飞世尔科技公司;Pfu DNA聚合酶(2.5 U/μL)、2×Basic Assemble Mix 北京全式金生物技术有限公司;细菌RNA提取试剂盒、cDNA第一链合成试剂盒、荧光定量PCR试剂盒、大肠杆菌感受态细胞DH5α 北京擎科生物科技有限公司;甲醇、酵母提取物、胰蛋白胨、琼脂粉、NaCl、MgSO4·7H2O、(NH4)2SO4、Na2HPO4·12H2O、KH2PO4、MnCl2·4H2O、ZnSO4·7H2O、CuSO4·5H2O、CaCl2、柠檬酸铁 分析纯,国药集团化学试剂有限公司;LaCl3·7H2O、庆大霉素(Gentamicin,Gm)、氨苄青霉素(Ampicillin,Amp) 分析纯,上海源叶生物科技有限公司;Tris-HCl 分析纯,北京酷莱博科技有限公司;LB培养基:蛋白胨10 g/L,酵母粉5 g/L,NaCl 10 g/L,121 ℃灭菌;MP基础培养基:MgSO4·7H2O 0.2 g/L,KH2PO4 1.4 g/L,(NH4)2SO4 3.0 g/L,Na2HPO4 3.0 g/L,CaCl2 30.0 mg/L,MnCl2·4H2O 5.0 mg/L,ZnSO4·7H2O 5.0 mg/L,CuSO4·5H2O 0.5 mg/L,柠檬酸铁30.0 mg/L,115 ℃灭菌,使用时添加甲醇至10 mL/L,得到MP培养基,在MP培养基中添加CaCl2或LaCl3的至终浓度为30 μmol/L,得到含各种金属离子的培养基。
A-14型常温高速离心机、1-15K型低温高速离心机 美国Sigma公司;9080型恒温培养箱 上海一恒科技公司;BP211D型分析天平(精度0.01 mg) 德国Sartorius公司;DYY-Ⅲ-8B型稳压稳流型电泳仪 北京六一仪器厂;WDP-160型低温振荡摇床 北京沃德创新医药科技中心;Gel-pro 2020D型凝胶成像仪 美国Pharmacia Biotech公司;FLC-3型超净台 哈尔滨东联有限公司;PRO-2612433型金属浴 北京金银杏生物科技有限公司;HZQ-F160型恒温振荡摇床 苏州培英实验设备有限公司;CFX96TM型荧光定量PCR仪、MiniPROTEAN®3 Cell型蛋白电泳仪 美国Bio-RAD 公司;T-1型PCR 仪 德国Biometra公司;UV-2100型紫外分光光度计 美国UNICO 公司;VC505型超声细胞破碎仪 美国Sonics 公司;Milropulser型电转化仪 北京友华照钦医疗器械有限公司。
1.2 实验方法
1.2.1 生物信息学分析
利用J1-1基因组序列和生物信息学分析筛选出基因组中甲醇脱氢酶基因,再通过BLAST与扭脱甲基杆菌AM1中的甲醇脱氢酶基因进行比对分析。基因和蛋白序列获得及序列比对分析网址:http//www.ncbi. nlm.nih.gov/,MP688登录号为CP002252.1,AM1登录号为CP001510.1。
1.2.2 分子生物学操作
基因组提取、质粒提取、PCR产物扩增、酶切、连接、转化等常规分子生物学操作参照文献[23]方法。
1.2.3 引物设计
文中所需引物序列如表1。
表 1 引物名称及序列Table 1. Names and sequences of primers引物名称 引物序列 UP-F CCGGTAAGGAGGAATTCTAACCGCCTTGATTCTTTCACGCATGC UP-R CCGTTTCCACGGTGTGCGTCCTTCGGAATCCTCCTCGTATGTTG Gm-F ATACGAGGAGGATTCCGAAGGACGCACACCGTGGAAACGG Gm-R GCCTAATCACAAGGCCTGTATTAGGTGGCGGTACTTGGGTCG DOWN-F ACCCAAGTACCGCCACCTAATACAGGCCTTGTGATTAGGCCGC DOWN-R GGTGGTGGTGCTCGAGTTTTTCAGGATCGCCCTGATAAATGGAACG 1.2.4 敲除菌J1-1Δmpq0771的构建
以J1-1基因组为模板,采用表1引物UP-F、UP-R、DOWN-F、DOWN-R分别扩增上、下游同源臂基因片段(1500 bp),引物Gm-F和Gm-R扩增Gm抗性基因片段(789 bp),产物经1%琼脂糖凝胶电泳后,用胶回收试剂盒纯化。pTIG质粒用EcoR I、Xho I内切酶进行双酶切,37 ℃反应2 h,产物经1%琼脂糖凝胶电泳回收。将上述回收产物利用2×Basic Assemble Mix重组试剂盒进行连接,获得的产物通过热激转化法转入E. coli DH5α感受态细胞中,转化后涂布至含有Amp抗性的LB平板上,37 ℃培养。挑取单克隆并活化,菌液PCR进行验证,结果为阳性的菌液进行测序鉴定。鉴定结果正确后,提取质粒pTIG-UP-Gm-DOWN,以该质粒为模板,使用引物UP-F、DOWN-R扩增片段UP-Gm-DOWN,片段经1%琼脂糖凝胶电泳回收,回收后电击转入J1-1感受态细胞中,通过Gm抗性筛选得到敲除菌J1-1Δmpq0771。
1.2.5 生长曲线及PQQ生物合成测定
将菌株接种至MP基础培养基中,30 ℃、200 r/min进行活化,活化后分别接种至含有不同金属离子的培养基中,每隔12 h测定OD600及PQQ合成水平,绘制生长曲线及PQQ合成曲线图,PQQ测定方法参考文献[24]中的光谱法。
1.2.6 甲醇脱氢酶酶活测定
取1 mL培养至对数中期的菌液,10000 r/min离心10 min后弃上清,重悬于1 mL 20 mmol/L的Tris-HCl(pH7.0)缓冲液中,置于冰浴中超声处理,4 ℃低温10000 r/min离心5 min,上清液用于酶活性检测,测定方法参考文献[20]。
1.2.7 甲醇脱氢酶蛋白表达测定
取1 mL已培养至对数中期的菌液,12000 r/min离心1 min后弃上清,重悬于500 μL 1×TE(10 mmol/L pH8.0 Tris-HCl)缓冲液中,置于冰上进行超声破碎(工作3 s,间歇3 s,2 min)。破碎后的菌液在12000 r/min下离心1 min,取上清进行十二烷基硫酸钠聚丙烯酰胺凝胶电泳(sodiumdodecyl sulfate polyacrylamide gel electrophoresis,SDS-PAGE)。
1.2.8 质谱鉴定
目的电泳条带切下装入无酶EP管中,封口膜封闭管口,送至北京华大基因有限公司进行蛋白质谱鉴定。
1.2.9 实时荧光定量PCR
将菌株J1-1分别接种至甲醇/无、甲醇/Ca2+、甲醇/La3+和甲醇/Ca2++La3+培养基中,培养至对数生长期,利用试剂盒提取RNA并逆转录为cDNA后进行荧光定量PCR,检测不同培养条件下mpq0771、mpq2496基因相对表达情况。
1.3 数据处理
各实验的指标测定均重复3次,结果用平均值±标准偏差表示,使用Microsoft Excel进行绘图;序列比对使用DNAMAN软件进行比对。
2. 结果与分析
2.1 菌株J1-1中甲醇脱氢酶基因的生物信息学分析
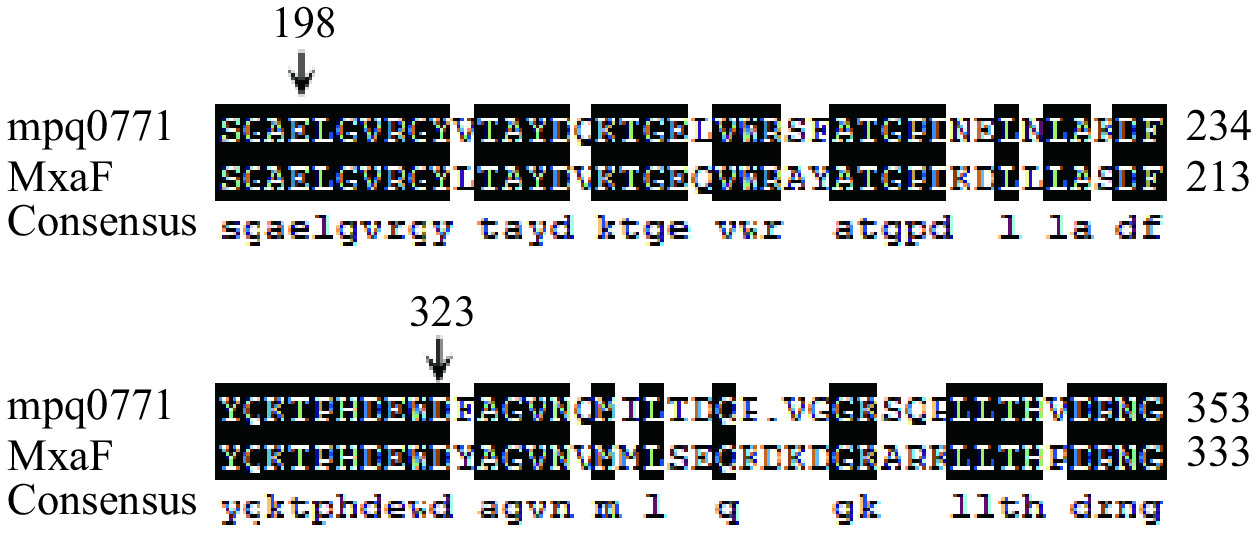
通过对J1-1全基因组序列系统的比对,J1-1中mpq0771基因与MxaF基因的核苷酸序列同源性为72%,氨基酸序列同源性为53%;mpq2496基因与XoxF基因的核苷酸序列同源性为66%,氨基酸序列同源性为53%;mpq0771基因与mpq2496基因的核苷酸序列同源性为61%,氨基酸序列同源性为48%。如图1所示,与MxaF型MDH的氨基酸序列的D303、E177对应mpq0771的D323、E198,推测为催化反应过程中所必需的,与Ca2+发生相互作用[25];如图2所示,与XoxF型MDH的氨基酸序列的D320对应mpq2496的D357,推测参与催化反应并与La3+进行配位[26]。综上结果提示,在菌株J1-1中可能也存在两种类型的甲醇脱氢酶。
2.2 Ca2+、La3+对菌株J1-1生长及PQQ生物合成的影响
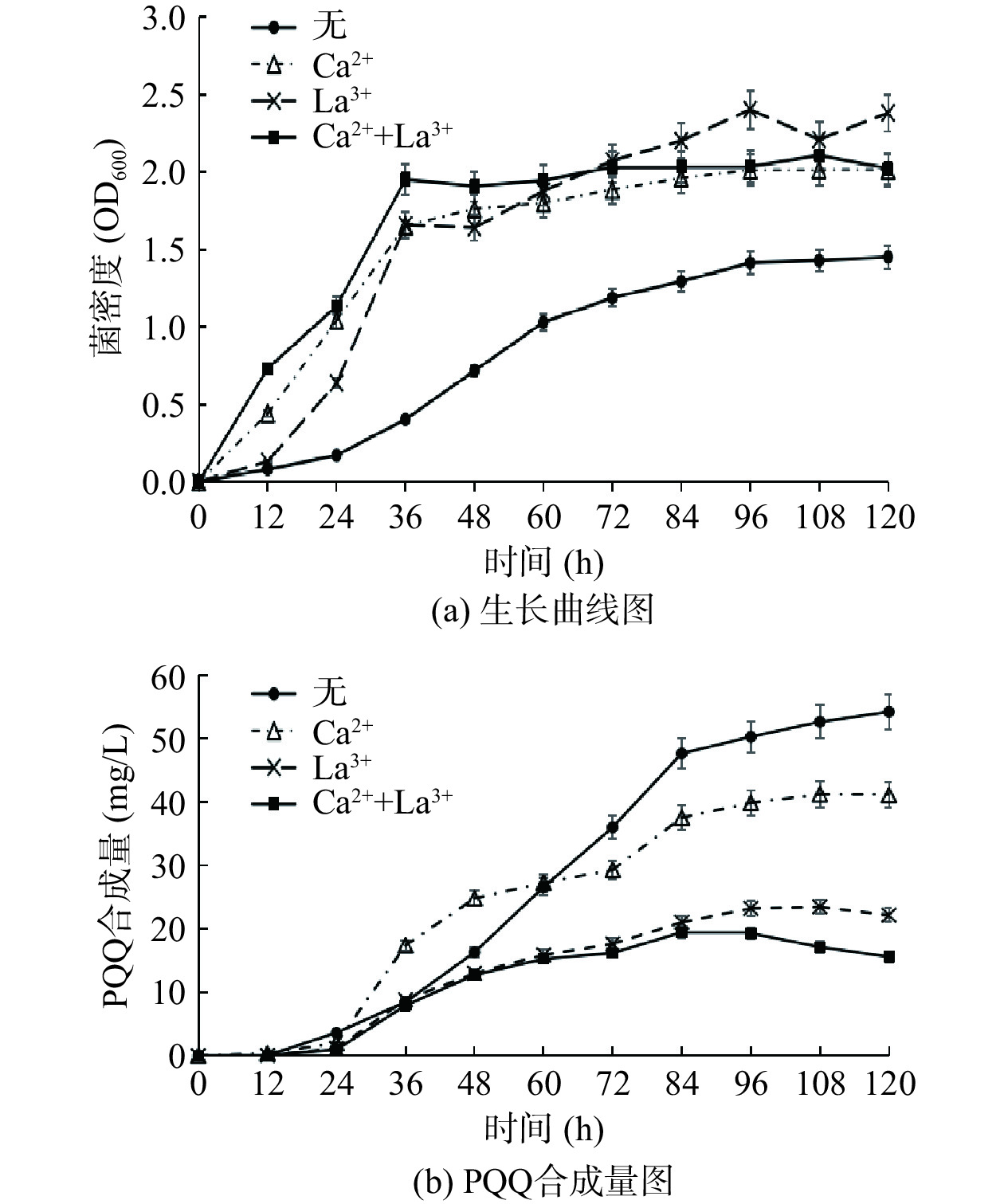
将菌株J1-1分别在甲醇/无、甲醇/Ca2+、甲醇/La3+和甲醇/Ca2++La3+ 4种培养基中培养,每隔12 h取样测定其OD600及PQQ产量并绘制曲线(如图3)。菌株J1-1在含有Ca2+、La3+的培养基中的生长速率均高于在无离子添加培养下的生长速率。前期时,菌株J1-1在添加Ca2+条件下的PQQ合成量高于野生菌,后期时合成量低于野生菌;菌株J1-1在两种含La3+培养基中的PQQ合成量一直低于野生菌。实验表明Ca2+、La3+均能够促进菌体的生长,但会显著降低PQQ合成量。
2.3 Ca2+、La3+对菌株J1-1甲醇脱氢酶活力的影响
取1 mL在甲醇/无、甲醇/Ca2+、甲醇/La3+和甲醇/Ca2++La3+ 4种培养基中的J1-1菌液,测定甲醇脱氢酶总酶活。如图4所示,在甲醇/La3+和甲醇/Ca2++La3+的培养条件下,菌株J1-1的甲醇脱氢酶活性高于甲醇/Ca2+的培养条件。前期研究表明,菌株J1-1的甲醇脱氢酶活性与PQQ产量呈负相关,MDH氧化甲醇速率加快,导致生成的甲醛累积,菌体“中毒”,从而PQQ产量下降[21],该实验结果与J1-1在甲醇/La3+和甲醇/Ca2++La3+中的PQQ合成量低于甲醇/Ca2+的现象相符。
2.4 Ca2+、La3+对菌株J1-1甲醇脱氢酶蛋白表达的影响
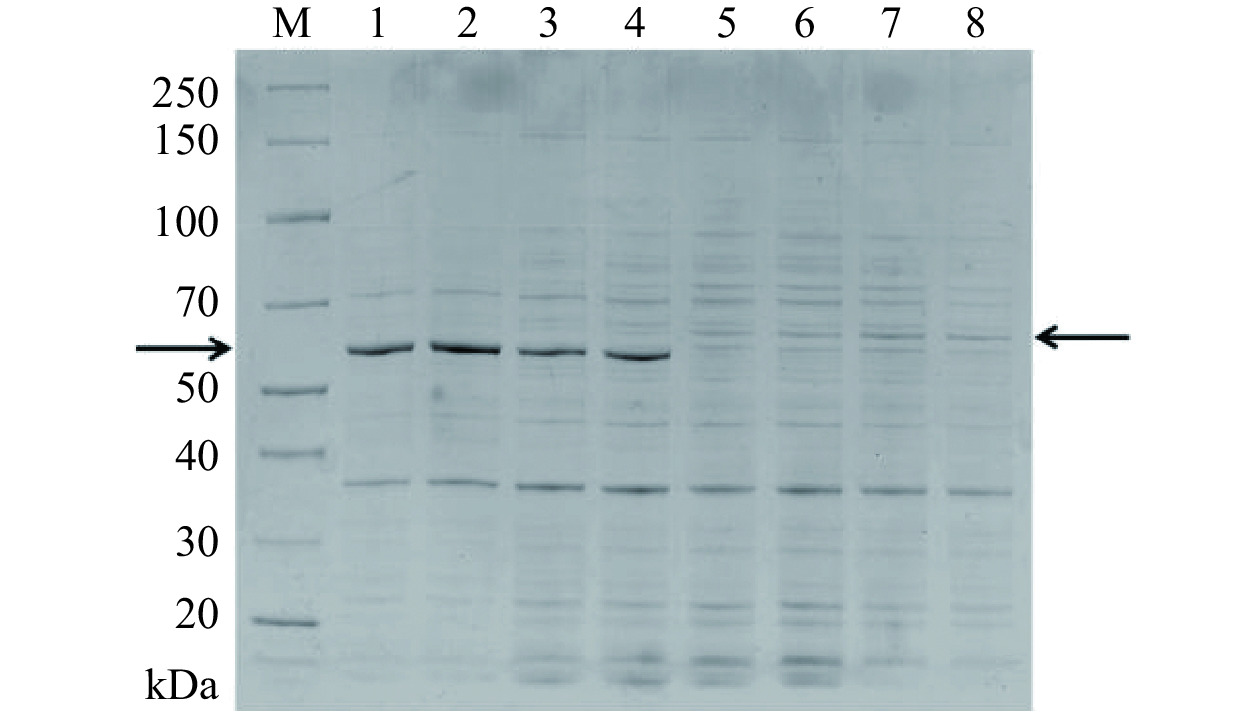
取1 mL在甲醇/无、甲醇/Ca2+、甲醇/La3+和甲醇/Ca2++La3+ 4种培养基中的J1-1菌液,进行超声处理,取全菌进行SDS-PAGE,蛋白电泳图谱如图5。根据前期实验结果可知,65 kDa处的蛋白条带为mpq0771基因编码的甲醇脱氢酶。mpq0771在甲醇/无(泳道1和2)、甲醇/Ca2+(泳道3和4)条件下有明显的表达;在甲醇/La3+(泳道5和6)和甲醇/Ca2++La3+(泳道7和8)的条件下无肉眼可见的条带,但在其上方处出现一条蛋白表达量增加的条带(蛋白分子量大约为68 kDa),胶内酶切并进行质谱分析鉴定。质谱结果如表2所示,该蛋白条带与mpq2496基因编码的甲醇脱氢酶匹配最佳,分值为4757,覆盖率为46%,且分子量大小也相符。以上结果显示mpq2496与文献[27−28]报道中XoxF的特征相似,La3+存在时XoxF基因表达更强。
表 2 质谱检测结果Table 2. Results of mass spectrum detection蛋白描述 分子量(Da) 分值 总肽段数 总序列数 丰度指数 覆盖率 PQQ依赖性脱氢酶 68 102 4754 231(183) 28(26) 5.57 46% TonB依赖性受体 68 330 3179 127(103) 20(20) 2.55 31% 甲酰甲基呋喃脱氢酶亚基α 62 050 1571 95(60) 19(18) 3.24 39% TonB依赖性受体塞 71 117 1243 76(49) 21(18) 1.70 36% 脯氨酰-tRNA合成酶 63 216 1018 69(39) 20(16) 1.75 39% 乙酰乳酸合成酶 64 977 949 66(39) 19(12) 1.68 26% 注:总肽段数:匹配到的总肽段数(高于显著性阈值的匹配数);总序列数:匹配到的总序列数(高于显著性阈值的序列数)。 2.5 Ca2+、La3+对菌株J1-1甲醇脱氢酶基因相对表达量的影响
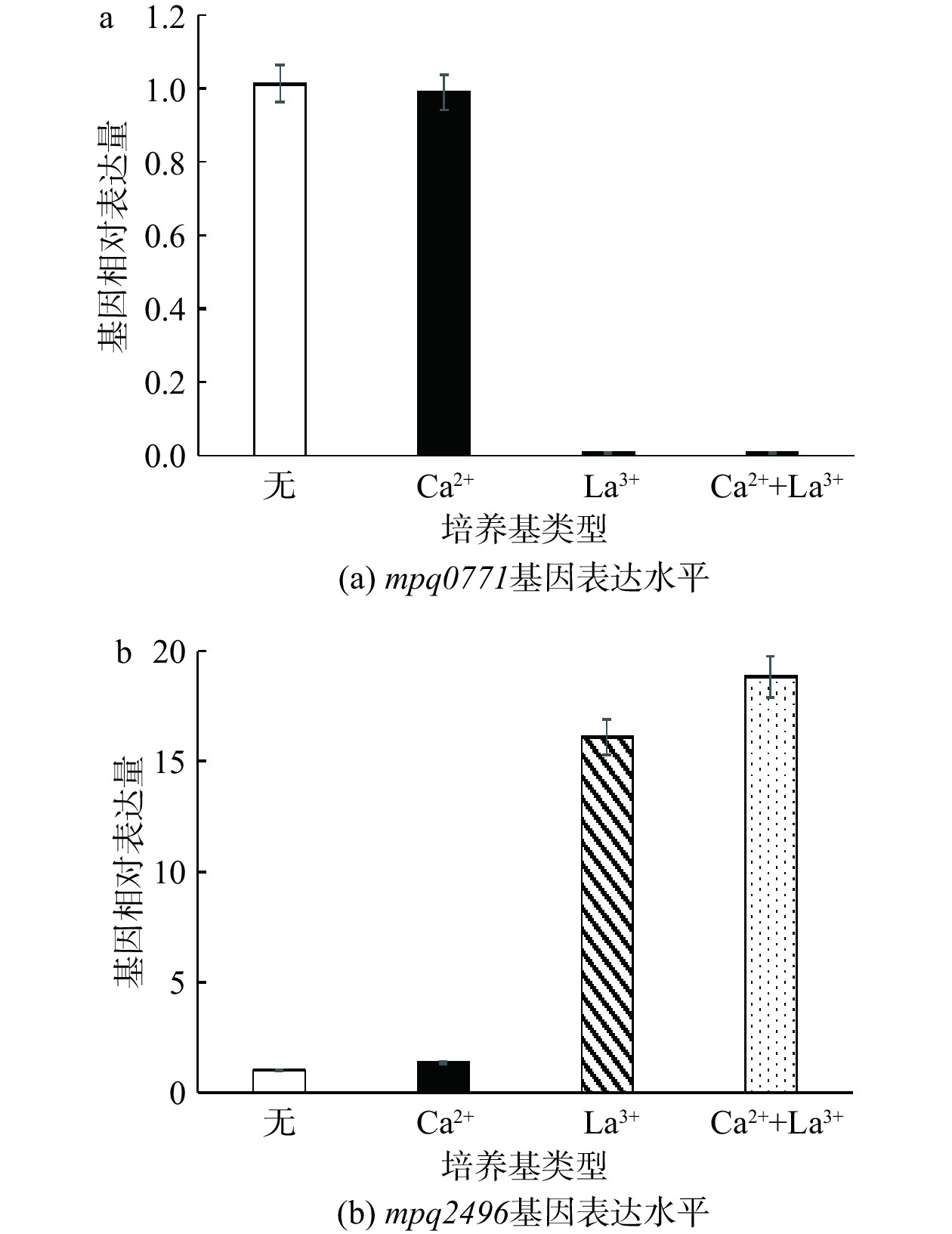
以cDNA为模板,GAPDH为内参基因,测定菌株J1-1在不同条件下mpq0771、mpq2496基因相对表达水平,结果分别如图6a和图6b所示。mpq0771和mpq2496参与甲醇代谢并发挥作用,且Ca2+、La3+对二者的表达水平均有影响。与甲醇/无条件相比,La3+存在时,mpq2496的表达水平均上调,mpq0771的表达水平均下调。与SDS-PAGE电泳结果相符,无离子添加和只添加Ca2+时,mpq0771表达水平较高,但在La3+存在时,mpq0771表达受到阻遏,mpq2496表达被激活。WANG等[29]研究也表明La3+可能会阻遏Ca2+依赖型酶和其他蛋白表达。
2.6 Ca2+、La3+对敲除菌J1-1Δmpq0771生长及PQQ生物合成的影响
mpq0771所编码的甲醇脱氢酶是菌株J1-1能够利用甲醇进行生长的关键基因,在甲醇代谢过程中发挥重要作用。如图7a所示,菌株J1-1在缺失mpq0771基因后,培养5 d后其菌密度仍低于0.2,不能利用甲醇生长。图7b为PQQ合成量图,结果显示J1-1Δmpq0771菌株可在含La3+的培养基中利用甲醇生长并能合成PQQ,说明有除mpq0771基因编码的甲醇脱氢酶在发挥作用,使菌株能够正常生长。
2.7 Ca2+、La3+对敲除菌J1-1Δmpq0771甲醇脱氢酶活力的影响
如图8所示,敲除菌J1-1Δmpq0771在Ca2+的培养基中不能利用甲醇进行生长,检测不到甲醇脱氢酶的活性;在甲醇/La3+和甲醇/Ca2++La3+条件下的甲醇脱氢酶酶活,敲除菌J1-1Δmpq0771可以利用甲醇且能检测到甲醇脱氢酶活性,说明mpq2496能够发挥甲醇脱氢酶的作用,也进一步反映了甲醇脱氢酶对于菌株利用和代谢甲醇的重要性。
2.8 Ca2+、La3+对敲除菌J1-1Δmpq0771甲醇脱氢酶蛋白表达的影响
敲除菌J1-1Δmpq0771的SDS-PAGE电泳结果如图9所示。J1-1Δmpq0771在甲醇为碳源的培养基中不能生长,但可在果糖为碳源的培养基中生长。在MP/果糖中J1-1Δmpq0771(泳道2)相较于野生菌J1-1(泳道1)的蛋白指纹图谱缺失一条65 kDa的甲醇脱氢酶蛋白条带,说明在J1-1Δmpq0771中mpq0771编码的甲醇脱氢酶不能正常表达,也印证了敲除基因mpq0771后菌株不能再利用甲醇。J1-1Δmpq0771菌株在La3+(泳道3)和Ca2++La3+(泳道4)的培养条件下,mpq2496编码的甲醇脱氢酶蛋白表达量较野生菌高,说明当La3+存在时,mpq2496能够代替mpq0771发挥甲醇脱氢酶的作用,即能够利用甲醇进行生长。
3. 结论
在菌株J1-1中存在着两种类型的甲醇脱氢酶,mpq0771基因编码的是Ca2+依赖的MxaF型MDH,mpq2496基因编码的是La3+依赖的XoxF型MDH,且均参与甲醇代谢。通过考察Ca2+、La3+对菌株J1-1生长状态、PQQ合成、MDH表达及酶活等的影响,发现敲除mpq0771基因会对菌株的生长及对甲醇的利用产生较大影响,提示该基因在甲醇代谢中的重要性,且当其表达量高时PQQ合成水平较高。研究还发现,La3+能使敲除菌J1-1Δmpq0771利用并代谢甲醇,该结果反映了La3+存在时mpq2496可以代替mpq0771发挥甲醇脱氢酶的作用,维持菌体生长,即La3+可使XoxF型MDH成为食甲基杆菌的主要甲醇脱氢酶[30],mpq0771与mpq2496之间还具有分工作用。
综上,菌株J1-1中mpq0771及mpq2496基因编码的甲醇脱氢酶在甲醇代谢中具有重要作用,也进一步说明了甲醇脱氢酶是甲醇代谢过程中的关键调控点[31]。菌株J1-1中的两种甲醇脱氢酶具有高表达的特性,提示可利用基因工程技术对此开发一种表达外源蛋白的系统。甲基营养菌具有特殊的一碳代谢网络,可实现利用生物转化生产高附加值产品,如化工产品、天然产物、食品添加剂等[32−33],因此对甲醇脱氢酶作用的研究不仅可为后续深入研究甲基营养菌甲醇利用途径提供理论依据,还有利于生物发酵业的发展。
-
表 1 引物名称及序列
Table 1 Names and sequences of primers
引物名称 引物序列 UP-F CCGGTAAGGAGGAATTCTAACCGCCTTGATTCTTTCACGCATGC UP-R CCGTTTCCACGGTGTGCGTCCTTCGGAATCCTCCTCGTATGTTG Gm-F ATACGAGGAGGATTCCGAAGGACGCACACCGTGGAAACGG Gm-R GCCTAATCACAAGGCCTGTATTAGGTGGCGGTACTTGGGTCG DOWN-F ACCCAAGTACCGCCACCTAATACAGGCCTTGTGATTAGGCCGC DOWN-R GGTGGTGGTGCTCGAGTTTTTCAGGATCGCCCTGATAAATGGAACG 表 2 质谱检测结果
Table 2 Results of mass spectrum detection
蛋白描述 分子量(Da) 分值 总肽段数 总序列数 丰度指数 覆盖率 PQQ依赖性脱氢酶 68 102 4754 231(183) 28(26) 5.57 46% TonB依赖性受体 68 330 3179 127(103) 20(20) 2.55 31% 甲酰甲基呋喃脱氢酶亚基α 62 050 1571 95(60) 19(18) 3.24 39% TonB依赖性受体塞 71 117 1243 76(49) 21(18) 1.70 36% 脯氨酰-tRNA合成酶 63 216 1018 69(39) 20(16) 1.75 39% 乙酰乳酸合成酶 64 977 949 66(39) 19(12) 1.68 26% 注:总肽段数:匹配到的总肽段数(高于显著性阈值的匹配数);总序列数:匹配到的总序列数(高于显著性阈值的序列数)。 -
[1] WANG C X, ZHANG B R, ZHAGN H Y, et al. Effect of dietary pyrroloquinoline quinone disodium in sows on intestinal health of the offspring[J]. Food & Function,2020,20(4):581−585.
[2] LIU L X, ZHAGN Y Y, LIU T, et al. Pyrroloquinoline quinone protects against exercise-induced fatigue and oxidative damage via improving mitochondrial function in mice[J]. The FASEB Journal,2021,35(4):21394.
[3] XU X, CHEN C, LU W J, et al. Pyrroloquinoline quinone can prevent chronic heart failure by regulating mitochondrial function[J]. Cardiovascular Diagnosis and Therapy,2020,10(3):453−469. doi: 10.21037/cdt-20-129
[4] YAMADA Y, NISHII K, KUWATA K, et al. Effects of pyrroloquinoline quinone and imidazole pyrroloquinoline on biological activities and neural functions[J]. Heliyon,2020,6(1):3240.
[5] 杨嵘, 张连成, 陶慧敏, 等. 吡咯喹啉醌对模拟急性高原暴露小鼠的抗缺氧和抗疲劳作用的研究[J]. 营养学报,2020,42(6):602−606. [YANG R, ZHANG L C, TAO H M, et al. Effects of pyrroloquinoline quinone on anti-hypoxia and anti-fatigue[J]. Acta Nutrimenta Sinica,2020,42(6):602−606.] YANG R, ZHANG L C, TAO H M, et al. Effects of pyrroloquinoline quinone on anti-hypoxia and anti-fatigue[J]. Acta Nutrimenta Sinica, 2020, 42(6): 602−606.
[6] 寇航, 王艳梅, 李彤, 等. 基于Methylovorus sp. J1-1基因组尺度代谢网络优化吡咯喹啉醌合成[J]. 生物技术通报,2022,38(2):173−183. [KOU H, WANG Y M, LI T, et al. Fermentation optimization for PQQ synthesis based on the genome-scale metabolic model of Methylovorus sp. J1-1[J]. Biotechnology Bulletin,2022,38(2):173−183.] KOU H, WANG Y M, LI T, et al. Fermentation optimization for PQQ synthesis based on the genome-scale metabolic model of Methylovorus sp. J1-1[J]. Biotechnology Bulletin, 2022, 38(2): 173−183.
[7] 张静, 刘孟粟, 秦志杰, 等. 吡咯喹啉醌高产菌株选育及发酵优化[J]. 食品与发酵工业,2022,48(16):56−64. [ZHANG J, LIU M S, QIN Z J, et al. Breeding and fermentation optimization of high titer pyrrorole-quinoline quinone strain[J]. Food and Fermentation Industries,2022,48(16):56−64.] ZHANG J, LIU M S, QIN Z J, et al. Breeding and fermentation optimization of high titer pyrrorole-quinoline quinone strain[J]. Food and Fermentation Industries, 2022, 48(16): 56−64.
[8] AKAGAWA M, MINEMATSU K, SHIBATA T, et al. Identification of lactate dehydrogenase as a mammalian pyrroloquinoline quinone (PQQ)-binding protein[J]. Scientific Reports,2016,6(1):26723. doi: 10.1038/srep26723
[9] JANJA T, KATARINA J, KAZUNOBU M. The highly tolerant acetic acid bacterium Gluconacetobacter europaeus adapts to the presence of acetic acid by changes in lipid composition, morphological properties and PQQ-dependent ADH expression[J]. Extremophiles,2007,11(4):627−635. doi: 10.1007/s00792-007-0077-y
[10] 孙青, 刘德华, 陈振. 甲醇的生物利用与转化[J]. 中国生物工程杂志,2020,40(10):65−75. [SUN Q, LIU D H, CHEN Z. Bioutilization and transformation of methanol[J]. China Biotechnology,2020,40(10):65−75.] SUN Q, LIU D H, CHEN Z. Bioutilization and transformation of methanol[J]. China Biotechnology, 2020, 40(10): 65−75.
[11] ANTHONT C, WILLIAMS P. The structure and mechanism of methanol dehydrogenase[J]. BBA-Proteins and Proteomics,2003,1647(1):18−23.
[12] RAN F. Preferential binding of lanthanides to methanol dehydrogenase evaluated with density functional theory[J]. The Journal of Physical Chemistry,2021,125(9):2251−2257. doi: 10.1021/acs.jpcb.0c11077
[13] 姚伦, 周雍进. 一碳化合物生物利用和转化研究进展[J]. 化工进展,2023,42(1):16−29. [YAO L, ZHOU Y J. Progress in microbial utilization of one-carbon feedstocks for biomanufacturing[J]. Chemical Industry and Engineering Progress,2023,42(1):16−29.] YAO L, ZHOU Y J. Progress in microbial utilization of one-carbon feedstocks for biomanufacturing[J]. Chemical Industry and Engineering Progress, 2023, 42(1): 16−29.
[14] 凡立稳, 王钰, 郑平, 等. 一碳代谢关键酶—甲醇脱氢酶的研究进展与展望[J]. 生物工程学报,2021,37(2):530−540. [FAN L W, WAGN Y, ZHEGN P, et al. Methanol dehydrogenase, a key enzyme of one-carbon metabolism:A review[J]. Chinese Journal of Biotechnology,2021,37(2):530−540.] FAN L W, WAGN Y, ZHEGN P, et al. Methanol dehydrogenase, a key enzyme of one-carbon metabolism: A review[J]. Chinese Journal of Biotechnology, 2021, 37(2): 530−540.
[15] WILLIAMS P A, COATES L, MOHAMMED F, et al. The atomic resolution structure of methanol dehydrogenase from Methylobacterium extorquens[J]. Acta Crystallographica. Section D, Biological Crystallography,2005,61:75−79. doi: 10.1107/S0907444904026964
[16] CHISTOSERDOVA L, LIDSTROM M E. Molecular and mutational analysis of a DNA region separating two methylotrophy gene clusters in Methylobacterium extorquens AM1[J]. Microbiology,1997,143:1729−1736. doi: 10.1099/00221287-143-5-1729
[17] HIBI Y, ASAI K, ARAFUKA H, et al. Molecular structure of La3+-induced methanol dehydrogenase-like protein in Methylobacterium radiotolerans[J]. Journal of Bioscience and Bioengineering,2010,111(5):547−549.
[18] NAKAGAWA T, MITSUI R, TANI A, et al. A catalytic role of XoxF1 as La3+-dependent methanol dehydrogenase in Methylobacterium extorquens strain AM1[J]. PLoS ONE,2017,7(11):50480.
[19] 邹琪琪, 齐姗姗, 谢录翰, 等. 细菌单杂交方法在筛选甲基营养菌甲醇脱氢酶启动子结合蛋白中的应用[J]. 浙江农业学报,2018,30(10):1705−1714. [ZOU Q Q, QI S S, XIE L H, et al. Application of bacterial-one-hybrid system in screening factors interating with methanol dehydrogenase gene promoter in a methylotrophic bacteria strain[J]. Acta Agriculturae Zhejiangensis,2018,30(10):1705−1714.] ZOU Q Q, QI S S, XIE L H, et al. Application of bacterial-one-hybrid system in screening factors interating with methanol dehydrogenase gene promoter in a methylotrophic bacteria strain[J]. Acta Agriculturae Zhejiangensis, 2018, 30(10): 1705−1714.
[20] 李淼鑫, 熊向华, 汪建华, 等. 甲基营养菌甲醇脱氢酶基因的克隆及表达[J]. 生物技术通讯,2010,21(6):779−782. [LI M X, XIONG X H, WAGN J H, et al. Cloning and expression of methanol dehydrogenase gene of a Methylotrophic bacteria strain[J]. Lett Biotechnol,2010,21(6):779−782.] LI M X, XIONG X H, WAGN J H, et al. Cloning and expression of methanol dehydrogenase gene of a Methylotrophic bacteria strain[J]. Lett Biotechnol, 2010, 21(6): 779−782.
[21] 孙晓宇, 薄明井, 杨亚欣, 等. 通过突变甲醇脱氢酶启动子提高甲基营养菌吡咯喹啉醌产量[J]. 生物技术通讯,2019,30(5):609−613,692. [SUN X Y, BO M J, YANG Y X, et al. Increasing pyrroloquinoline quinone yield of Methylovorus sp. by methanol dehydrogenase promoter mutation[J]. Letters in Biotechnology,2019,30(5):609−613,692.] SUN X Y, BO M J, YANG Y X, et al. Increasing pyrroloquinoline quinone yield of Methylovorus sp. by methanol dehydrogenase promoter mutation[J]. Letters in Biotechnology, 2019, 30(5): 609−613,692.
[22] 李大攀, 葛欣, 魏静远, 等. 甲基营养菌MP688甲醇脱氢酶基因mpq1818的敲除及功能研究[J]. 生物技术通讯,2014,25(5):632−635. [LI D P, GE X, WEI J Y, et al. Knockout and characterization of mpq1818 gene of Methylovorus sp. MP688[J]. Letters in Biotechnology,2014,25(5):632−635.] LI D P, GE X, WEI J Y, et al. Knockout and characterization of mpq1818 gene of Methylovorus sp. MP688[J]. Letters in Biotechnology, 2014, 25(5): 632−635.
[23] 奥斯博. 精编分子生物学实验指南[M]. 第4版. 北京:科学出版社, 1998:25−127,652. [AUSUBEL F M. Short protocols in molecular biology[M]. 4th ed. Beijing:Science Press, 1998:25−127,652.] AUSUBEL F M. Short protocols in molecular biology[M]. 4th ed. Beijing: Science Press, 1998: 25−127,652.
[24] 杨延新, 熊向华, 游松, 等. 3种检测吡咯喹啉醌的方法比较[J]. 生物技术通讯,2011,22(4):544−547. [YANG Y X, XIONG X H, YOU S, et al. Comparing three kinds of pyrroloquinoline quinone detection methods[J]. Letters in Biotechnology,2011,22(4):544−547.] YANG Y X, XIONG X H, YOU S, et al. Comparing three kinds of pyrroloquinoline quinone detection methods[J]. Letters in Biotechnology, 2011, 22(4): 544−547.
[25] 董方, 张惟材, 汪建华, 等. 醌蛋白研究进展[J]. 生物技术通讯,2007,18(1):132−136. [DONG F, ZHANG W C, WANG J H, et al. Advances in quinoprotein research[J]. Letters in Biotechnology,2007,18(1):132−136.] DONG F, ZHANG W C, WANG J H, et al. Advances in quinoprotein research[J]. Letters in Biotechnology, 2007, 18(1): 132−136.
[26] NATHAN M G, MATTHIAS F, KEMAL D, et al. Lanthanide-dependent alcohol dehydrogenases require an essential aspartate residue for metal coordination and enzymatic function[J]. The Journal of Biological Chemistry,2020,295(24):8272−8284. doi: 10.1074/jbc.RA120.013227
[27] PAULA R J, TOMASZ K, ROBERT S, et al. Occurrence of XoxF-type methanol dehydrogenases in bacteria inhabiting light lanthanide-rich shale rock[J]. FEMS Microbiology Ecology,2021,97(2):259. doi: 10.1093/femsec/fiaa259
[28] CHU F, LIDSTROM M E. XoxF acts as the predominant methanol dehydrogenase in the type I Methanotroph Methylomicrobium buryatense[J]. Journal of Bacteriology,2016,198(8):1317−1325. doi: 10.1128/JB.00959-15
[29] WANG L H, ZHOU Q, LU T H, et al. Molecular and cellular mechanism of the effect of La(III) on horseradish peroxidase[J]. Journal of Biological Inorganic Chemistry,2010,15(7):1063−1069. doi: 10.1007/s00775-010-0665-7
[30] TANI A, MITSUI R, NAKAGAWA T. Discovery of lanthanide-dependent methylotrophy and screening methods for lanthanide-dependent methylotrophs[J]. Methods in Enzymology,2021,650:11−18.
[31] 周狄霏, 冯晨曦, 宋书真, 等. 甲基营养菌代谢过程新进展与代谢工程改造[J]. 南京工业大学学报(自然科学版),2022,44(5):511−522. [ZHOU D F, FENG C X, SONG S Z, et al. New advances in metabolic process and metabolic enineering in methylotrophs[J]. Journal of Nanjing Tech University (Natural Science Edition),2022,44(5):511−522.] ZHOU D F, FENG C X, SONG S Z, et al. New advances in metabolic process and metabolic enineering in methylotrophs[J]. Journal of Nanjing Tech University (Natural Science Edition), 2022, 44(5): 511−522.
[32] CHISTOSERDOVA L. Modularity of methylotrophy, revisited[J]. Environmental Microbiology,2011,13(10):2603−2622. doi: 10.1111/j.1462-2920.2011.02464.x
[33] 郭姝媛, 吴良焕, 刘香健, 等. 微生物中一碳代谢网络构建的进展与挑战[J]. 合成生物学,2022,3(1):116−137. [GUO S Y, WU L H, LIU X J, et al. Developing C1-based metabolic network in methylotrophy for biotransformation[J]. Synthetic Biology Journal,2022,3(1):116−137.] GUO S Y, WU L H, LIU X J, et al. Developing C1-based metabolic network in methylotrophy for biotransformation[J]. Synthetic Biology Journal, 2022, 3(1): 116−137.





 下载:
下载:
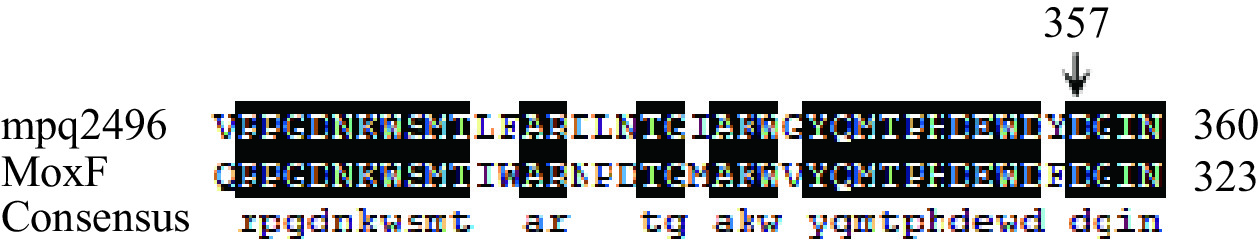




 下载:
下载:
