Bacterial Community Analysis of Shanxi Aged Vinegar during the Fermentation by High-throughput Sequencing
-
摘要: 以山西老陈醋醋醅为研究对象,通过高通量测序技术,探究其在发酵过程中细菌群落的多样性和演替规律。结果表明,山西老陈醋醋酸发酵过程中在门的水平上主要有厚壁菌门(Firmicutes)、变形菌门( Proteobacteria)、放线菌门(Actinobacteria)和拟杆菌门(Bacteroidetes)等7个菌门,其中厚壁菌门的相对丰度随发酵进行呈上升趋势,至发酵末期,其相对丰度甚至达到98%以上,在整个醋酸发酵阶段处于主导地位。在属水平,参与山西老陈醋醋酸发酵阶段的主要细菌有乳杆菌属(Lactobacillus,57.4%~66.4%)、醋酸菌属(Acetobacter,3.8%~27.3%)、链球菌属(Streptococcus,0.3%~3.0%)、魏斯氏菌属(Weissella,0.2%~1.8%)、芽孢杆菌属(Bacillus,0.1%~1.2%)、假单胞菌属(Pseudomonas,0%~1.8%)等,其中乳杆菌属(Lactobacillus)和醋酸菌属(Acetobacter)的细菌占主导优势,发酵前5 d二者的相对丰度之和高达90%左右。发酵前期的醋醅样品中并未检测到假单胞菌属(Pseudomonas),随着醋酸发酵的进行其在第5 d的样本中出现,并在第7 d达到峰值(1.1%~3.4%)。山西老陈醋醋酸发酵阶段采用“套醪接种”,不同发酵批次的细菌群落结构和变化情况基本相同,所共有的微生物物种占比总数的70%以上,说明山西老陈醋固态酿造工艺的稳定性。Abstract: By high-throughput sequence methods, the research explored the diversity and succession of the bacterial communities in fermented grains of Shanxi aged vinegar. The results showed that during the acetic acid fermentation of Shanxi aged vinegar, mainly 7 phyla were at the phylum level, including Firmicutes, Proteobacteria, Actinobacteria, and Bacteroidetes, among which the relative abundance of Firmicutes showed an increasing trend during the fermentation, and finally even reached over 98%, dominating the entire fermentation stage. At the genus level, the main bacteria participating in the acetic acid fermentation stage of Shanxi aged vinegar included Lactobacillus (57.4%~66.4%), Acetobacter (3.8%~27.3%), Streptococcus (0.3%~3.0%), Weissella (0.2%~1.8%), Bacillus (0.1%~1.2%), Pseudomonas (0%~1.8%), etc, among which Lactobacillus and Acetobacter bacteria dominated, with a relative abundance of around 90% in the first 5 days of fermentation. Meanwhile, Pseudomonas was not detected in the vinegar Pei samples during the early stage of the acetic acid fermentation, but appeared in the samples on the 5th day and reached its peak (1.1%~3.4%) on the 7th day. Besides, "mash inoculation" adopted during the acetic acid fermentation of Shanxi aged vinegar, the bacterial community structure as well as changes in different fermentation batches were basically the same, and the shared microbial species accounted for more than 70% of the total, indicating the stability of the solid-state fermentation process of Shanxi aged vinegar.
-
山西老陈醋是我国北方最具代表性的食醋,其酿造工艺主要包括:制曲、酒精发酵(AF)、醋酸发酵(AAF)、熏醅及陈酿等[1−2],并于2006年荣获首批“国家非物质文化遗产”称号[3]。山西老陈醋醋酸发酵过程采用的是固态发酵工艺,依靠酒醪、辅料(稻壳、麸皮、谷糠)及空气中的微生物自发富集性,在发酵剂“种醅”的作用下启动发酵,酿造过程中复杂的微生物群落及其代谢产物赋予了传统食醋独特的风味[4]。随着醋酸发酵的进行,高温、高酸使得耐受性差的微生物逐渐消亡,适应该环境的微生物通过增殖、代谢成为优势菌,从而完成发酵基质自身“净化”,抑制杂菌滋生,保障醋酸发酵顺利、平稳进行。优势微生物的种类及其代谢生成的产物是影响山西老陈醋品质的重要因素,参与发酵的核心微生物的菌群结构及演替规律也是解析山西老陈醋酿造机理的关键[5]。
随着科学技术的飞速发展,免培养的分子生态学技术如变性梯度凝胶电泳(PCR-DGGE)[6−8]、高通量测序[9−11]、宏基因组学[12−13]等在近年来也被广泛地应用于传统食醋酿造过程中微生物多样性的研究。范三红等[14]利用高通量测序对山西陈醋发酵过程中的微生物群落组成进行了探讨,结果表明山西陈醋醋曲、酒精发酵阶段和醋酸发酵阶段的微生物群落结构具有差异性,并随着发酵进行不断变化;杨玲等[15]在研究山西老陈醋和怀仁醋酒精发酵阶段细菌菌群多样性时发现,山西老陈醋样品的细菌菌群多样性更高,两种工艺酒精发酵阶段样品细菌种类存在显著差异;Zhu等[16]研究了山西老陈醋醋酸发酵阶段微生物的群落结构和代谢产物;Nie等[17]系统地研究了山西老陈醋大曲、酒精发酵及醋酸发酵阶段细菌和真菌的群落结构,并进行了微生物群落演替与代谢产物的相关性分析。上述研究为科学、合理地解析山西老陈醋酿造过程中微生物群落变化提供了重要的理论基础,但并未对不同批次的菌群结构进行深入研究,而参与发酵微生物的种类及其代谢产物与产品质量息息相关。
山西老陈醋的接种采用“套醪接种”的方式进行,即将成熟的酒精醪液和麸皮、稻壳、谷糠充分混匀之后,取品温39~42 ℃的微生物代谢最为旺盛的优良醋醅(上一批次)作为种醅,按10%的量接种至新鲜入缸/池的醋醅中,在醋酸发酵过程中不再接种任何外源的微生物。本研究跟踪采集了3个连续批次的山西老陈醋醋醅样本,通过高通量测序技术研究醋酸发酵阶段细菌的群落结构和演替规律,重在探索不同批次食醋品质保持稳定性的内在因素,为山西老陈醋采用“套醪接种”提供科学依据的同时,揭示醋酸发酵过程中优势物种及低丰度菌群生物量的动态变化,便于深入了解山西老陈醋的核心微生物并进行过程控制。
1. 材料与方法
1.1 材料与仪器
老陈醋 采集地点:2022年5月~6月,山西老陈醋集团有限公司醋酸发酵车间(山西,晋中);QIAamp DNA Microbiome Kit 德国QIAGEN;琼脂糖 生工生物工程(上海)股份有限公司;TE 缓冲液(pH8.0) 北京博美富鑫科技有限公司;EB(红色荧光核酸染料) 青岛拓邦生物科技有限公司;Tris base、乙酸(冰醋酸) 北京博美富鑫科技有限公司;EDTA二钠 北京博美富鑫科技有限公司;100 bp DNA Ladder Marker 北京六合通经贸有限公司;无水乙醇 青岛拓邦生物科技有限公司;VAHTSTM DNA Clean Beads 南京诺唯赞生物科技有限公司。
Dy NA Quant 200浓度测定仪 美国Pharmacia Biotech;DYY-6C型琼脂糖凝胶电泳仪 北京市六一仪器厂;Chemidoc-It 810凝胶成像系统 英国UVP;MINI-smart centrifuge小型离心机 昊诺斯生物;Centrifuge5424高速冷冻离心机 德国Eppendorf;FA224电子天平 上海舜宇恒平科学仪器有限公司;7500实时荧光定量PCR仪 美国ABI;序列测定 深圳华大基因股份有限公司。
1.2 实验方法
1.2.1 醋醅样品的采集方法
a. 采样设计:随机跟踪连续的3个醋酸发酵正常批次(标记为A、B、C)进行取样,发酵周期9 d,隔天在翻醅后取样(分别标记为A1,A3,A5,A7,A9;B1,…,B9;C1,…,C9),最终样本总计15个(表1)。
表 1 山西老陈醋不同批次醋醅取样时间及其编号Table 1. Sampling times and batch numbers of Pei samples during acetic acid fermentation (AAF) of Shanxi aged vinegar批次 取样时间 Day 1 Day 3 Day 5 Day 7 Day 9 混合 #1 A1 A3 A5 A7 A9 A #2 B1 B3 B5 B7 B9 B #3 C1 C3 C5 C7 C9 C 混合 AAF1 AAF3 AAF5 AAF7 AAF9 D b. 采样方法:在发酵缸四个顶点以及中心位置,采集距离醋酸发酵缸表面20 cm处的醋醅并充分混匀,然后通过四分法舍去多余的醋醅,最终将300~500 g醋醅样品收集于已灭菌的自封袋中,冷藏带回实验室后放置于−70 ℃冰箱中保存。
1.2.2 DNA的提取
对上述15个醋醅样本进行DNA的提取,使用QIAamp DNA Microbiome Kit试剂盒,按照说明书提取。提取的DNA通过Dy NA Quant 200浓度测定仪及1%的琼脂糖凝胶进行浓度和质量检测。质检合格的样本置于−20 ℃冰箱中保存以备后续实验使用,不合格的样本重新提取DNA。
1.2.3 PCR扩增和测序
采用细菌16S rDNA V3~V4扩增通用引物:338F(ACTCCTACGGGAGGCAGCAG)and 806R(GGACTACHVGGGTWTCTAAT)[18]。PCR的扩增体系为10 μL:基因组DNA 50 ng、引物(10 μmol/L)F和R各0.3 μL、5 μL KOD FX Neo Buffer、2 μL dNTP、0.2 μL KOD FX Neo,加ddH2O定容到10 μL。
PCR反应条件:95 ℃预变性5 min;95 ℃变性30 s,50 ℃退火30 s,72 ℃延伸30 s,完成一个循环,循环共重复25次;72 ℃最终延伸7 min,4 ℃保存[19]。对符合要求的产物进行后续实验。
在Illumina MiSeq平台(Illumina Inc.,San Diego,CA,USA)上进行Paired-end测序。
1.3 数据处理
15个醋醅样本各测定一次,测序完成后,进行数据分析和序列优化,首先去除下机数据中的低质量reads。序列拼接使用软件FLASH[20](Fast Length Adjustment of Short reads,v1.2.11),利用重叠关系将双末端测序得到的成对reads组装成一条序列,从而得到高变区的Tags。利用软件USEARCH(v7.0.1090)[21]将拼接好的Tags,在97%的相似度下进行聚类,得到OTU(Operational Taxonomic Units)的代表序列。随后,通过RDP classifer(v2.2)软件将OTU代表序列与数据库(Greengene V201305)比对进行物种注释,置信度阈值设置为0.60。使用R(v3.1.1)软件中的gplots包进行物种聚类热图分析,距离算法为euclidean,聚类方法为complete。利用Alpha多样性指数绘制盒形图,差异分析与作图均通过R软件(v3.1.1)进行,并通过R(v3.1.1)软件中ade4包进行PCA 3D主成分分析与作图。
2. 结果与分析
2.1 醋醅样本中细菌群落测序分析
通过Illumina MiSeq对15个醋醅样本进行测序,共获得1324100条目标序列(Tags),每个样品净Tags约为88273,Tags平均长度为252 bp。经过质控筛选后,在97%的相似度下,将其聚类为用于物种分类的操作分类单元(OTU)(表2)。OTU的丰度初步说明了样品的物种丰富程度。随着醋酸发酵的进行,#1、#2、#3批次的OTU数量先增加后减少,并在第5 d达到最大值(约95个OTU)。
表 2 各样本中细菌群落测序情况以及在97%相似度下OTU划分情况Table 2. Sequencing of bacterial communities and OTU division under 97% similarity in each Pei sample样品名称 净序列 平均长度(bp) 有效序列(%) 序列数量 OTU数量(个) A1 88805 252 99.1 81013 78 A3 87778 252 99.27 82005 83 A5 88289 252 99.26 81198 95 A7 89346 252 99.11 84549 89 A9 87294 252 98.9 85046 71 B1 87886 252 98.99 81733 81 B3 88624 252 98.87 83436 80 B5 87933 252 99.12 82185 94 B7 88474 252 98.89 81332 95 B9 87894 252 98.63 85801 72 C1 87898 252 98.95 81356 86 C3 88418 252 98.96 80735 87 C5 88911 252 99.3 81611 98 C7 89143 252 99.22 83491 94 C9 87407 252 99.2 85763 66 2.2 OTU Venn图分析
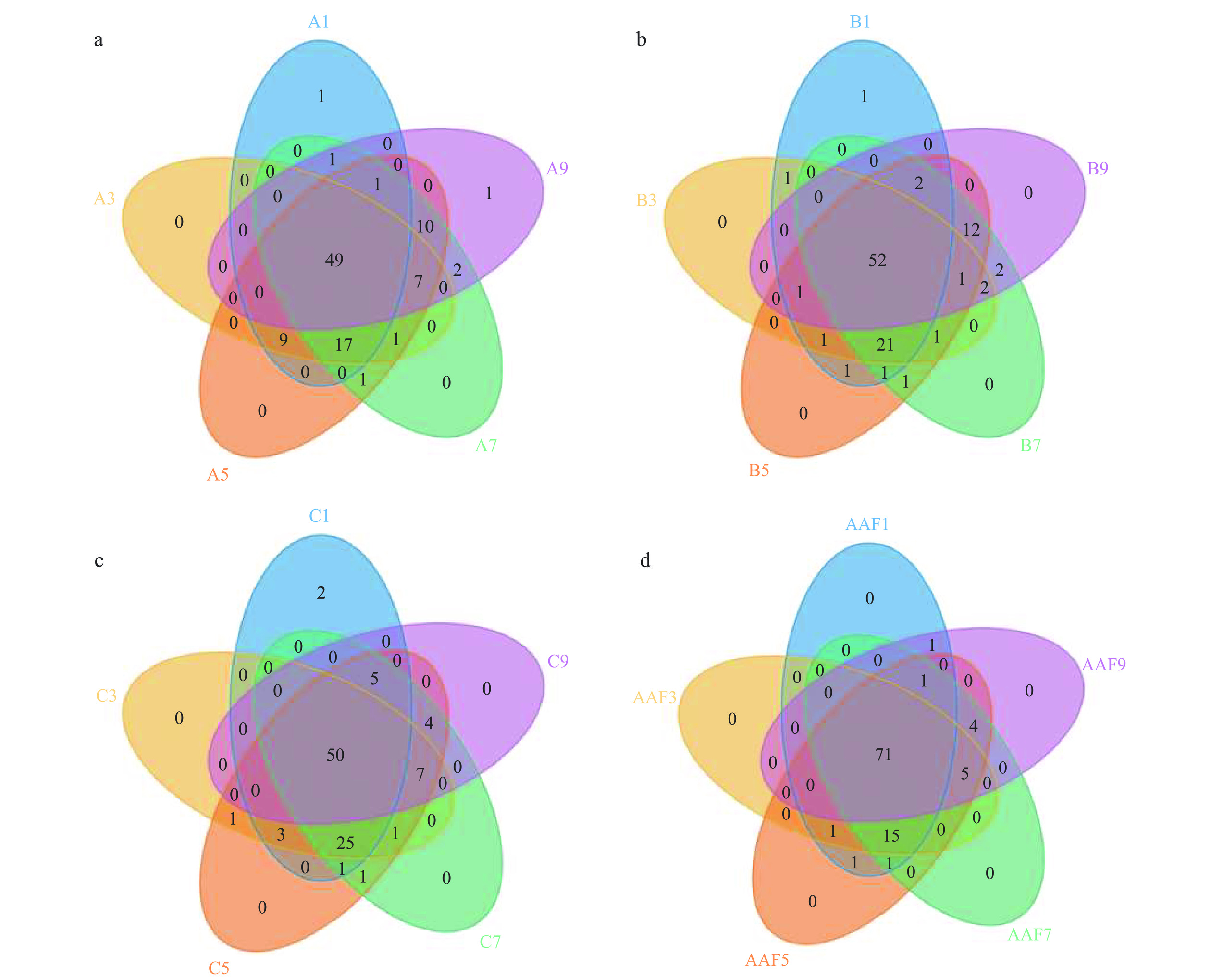
Venn图可以直观展示多个样品共有的和各自特有的OTU数目,显示样本的OTU数目组成相似性及重叠情况[22]。不同颜色重叠部分数字是两个及两个以上样品共有的OTU个数,非重叠部分是各样品所特有的OTU。根据OTU所代表的物种即可以知道不同环境中的核心微生物种类。图1为各批次醋酸发酵阶段OTU 的Venn图结果。
![]() 图 1 各批次醋酸发酵阶段OTU的Venn图注:图中a、b、c代表三个发酵批次,d为三个批次的混合物。同样颜色代表不同发酵批次相同发酵时间的醋醅(如:A1、 B1、 C1)。A1、A3、…、A9表示同一发酵批次1、3、…、9 d的醋醅样本;AAF是将三个批次相同发酵时间的醋醅混合在一起;图2同。Figure 1. Venn diagram of OTU in each batch of acetic acid fermentation stage
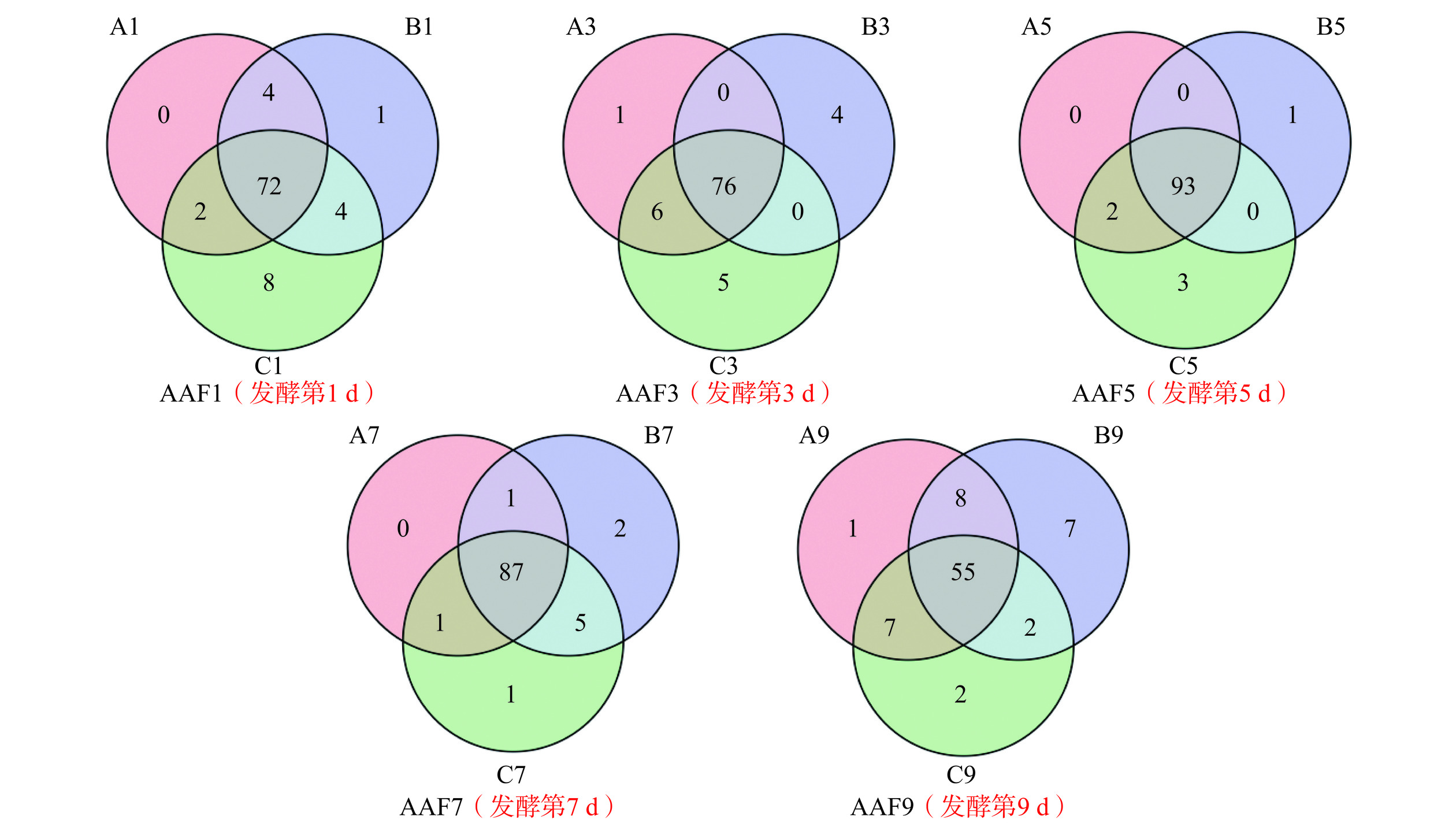
图 1 各批次醋酸发酵阶段OTU的Venn图注:图中a、b、c代表三个发酵批次,d为三个批次的混合物。同样颜色代表不同发酵批次相同发酵时间的醋醅(如:A1、 B1、 C1)。A1、A3、…、A9表示同一发酵批次1、3、…、9 d的醋醅样本;AAF是将三个批次相同发酵时间的醋醅混合在一起;图2同。Figure 1. Venn diagram of OTU in each batch of acetic acid fermentation stage山西老陈醋发酵过程中存在着复杂的微生物群落,随着发酵的进行,醋醅中的微生物也在不断的消亡、增殖、演替。从图1 a~c可以看出,#1批次五个样品共有的OTU个数为49个,占总OTU的49%;#2批次五个样品中共有的OTU个数为52个,占总OTU的52%;#3批次五个样品中共有的OTU个数为50个,占总OTU的50%。当三个批次的15个样品汇集在一起时,在整个发酵过程中检测到共有OTU占总OTU的71%(图1d)。固态发酵基质的复杂性和分层性导致细菌群落的丰度及多样性一直处于与外界环境的调整与适应中,经过几百年的驯化和演变,逐渐形成相对稳定的、适应山西老陈醋发酵环境的群落结构,而这些微生物也是促使发酵顺利进行、保证不同批次产品质量统一的关键所在。
不同批次、同一发酵时间的醋醅样品中共有OTU个数不尽相同,但共有OTU数目远大于各样品特有OTU的数目,这很大程度上与山西老陈醋的“接种”工艺息息相关,即将上一批发酵第2 d或第3 d的醋醅作为种醅,接种于下一批拌好辅料的新醅中。图2表明,不同批次的醋酸发酵过程中细菌群落结构及其变化情况基本相同,相同发酵时间的醋醅样品中细菌物种具有很大的相似性,这说明醋酸发酵过程中细菌菌群是相对稳定的,这为最终产品的质量稳定提供了保证。特有OUT的出现,推测可能与原料的产地、成分、附着的微生物类型,发酵车间的温、湿度变化有关系,但还需进一步验证。
2.3 醋醅样本中细菌群落Alpha多样性分析
Alpha多样性主要是分析样品中微生物群落的多样性和丰度,包括observed species、Chao、Ace,Shannon以及Simpson等指数[23]。通常,前面4个指数越大,最后一个指数越小,说明样品中的物种越丰富。其中,observed species指数、Chao指数和Ace指数反映样品中群落的丰富度,即简单指群落中物种的数量,而不考虑群落中每个物种的丰度情况。而Shannon指数以及Simpson指数反映群落的多样性,受样品群落中物种丰富度和物种均匀度的影响。相同物种丰富度的情况下,群落中各物种具有越大的均匀度,则认为群落具有越大的多样性。Coverage用于表征各醋醅样本文库的覆盖率,反映测序深度是否能覆盖整个微生物群落,是否能代表样本中所有微生物的真实情况[24]。
基于本课题组前期研究发现,前5 d微生物的碳源主要为乙醇,从第7 d开始醋醅里的酒精几乎消耗殆尽,同时醋醅的温度从第7 d(44.6 ℃)开始大幅下降,至发酵结束时(9 d)降至35.5 ℃,因此将发酵前5 d划分为前中期,第7~9 d列为发酵后期。从表3中可以看到,醋酸发酵过程中醋醅细菌的菌群丰富度和多样性随着发酵的进行不断变化。各批次醋酸发酵的前中期,细菌菌群丰富度和多样性明显高于发酵后期的样本,说明发酵前中期细菌种类较为丰富,除了种醅所带入的微生物外,还有很大一部分来源于辅料、酒醪以及空气。
表 3 醋醅样本中细菌群落的Alpha多样性Table 3. Alpha diversity of bacterial community in Pei samples样品名称 Sobs Chao指数 Ace指数 Shannon指数 Simpson指数 Coverage A1 86 77.60 76.88 2.14 0.18 99% A3 73 80.00 76.21 2.06 0.20 99% A5 76 86.00 86.18 2.26 0.16 99% A7 72 78.10 79.58 1.49 0.40 99% A9 69 92.00 93.70 0.64 0.79 99% B1 80 79.43 79.81 1.86 0.26 99% B3 75 103.25 101.24 2.14 0.18 99% B5 77 86.20 103.37 2.03 0.24 99% B7 73 80.17 80.72 2.20 0.17 99% B9 68 82.09 83.08 0.62 0.79 99% C1 85 96.00 87.23 1.89 0.24 99% C3 80 83.00 81.58 2.21 0.17 99% C5 72 100.17 97.76 2.26 0.15 99% C7 72 93.20 87.57 1.66 0.36 99% C9 61 69.67 71.82 0.47 0.84 99% 随着醋酸发酵过程的推进,细菌菌群的丰度、多样性迅速下降,大多数的外源微生物因不能适应高温和酸性酿造环境从而消失或维持低生命活性状态,而环境耐受性强的细菌经过短暂的延滞期后,开始以乙醇为碳源大量繁殖,产生初级代谢物和次级代谢物。发酵中期,细菌群落的丰富度和多样性有增加趋势,推测可能有以下几个原因:延滞期较长或处于休眠状态的微生物,在某些代谢产物(糖、肽、氨基酸)的刺激下逐渐开始活动;发酵中期环境中的乙醇浓度已大大下降,受乙醇抑制的微生物也开始增殖[25]。从发酵第7 d开始,细菌的丰富度和多样性又有较小幅度下降,很大原因是微生物由于营养匮乏而进入了衰亡期。随着测序深度的增加,多样性指数的增加趋势也逐渐变缓,且最终达到了饱和。每个醋醅样本的测序量(Coverage)均达到99%以上,说明目前的测序量是合理的,醋醅中细菌群落的多样性基本都能检测到。
2.4 醋醅样品的细菌丰度及多样性分析
通过与数据库对比进行样品中OTU的物种分类,分别在门和属2个分类等级,绘制醋醅样品的物种profiling面积图和柱状图。图3~图4直观地展示了不同物种在每个样品中所占的比例。
2.4.1 醋酸发酵阶段细菌群落在门水平的结构分布以及动态变化
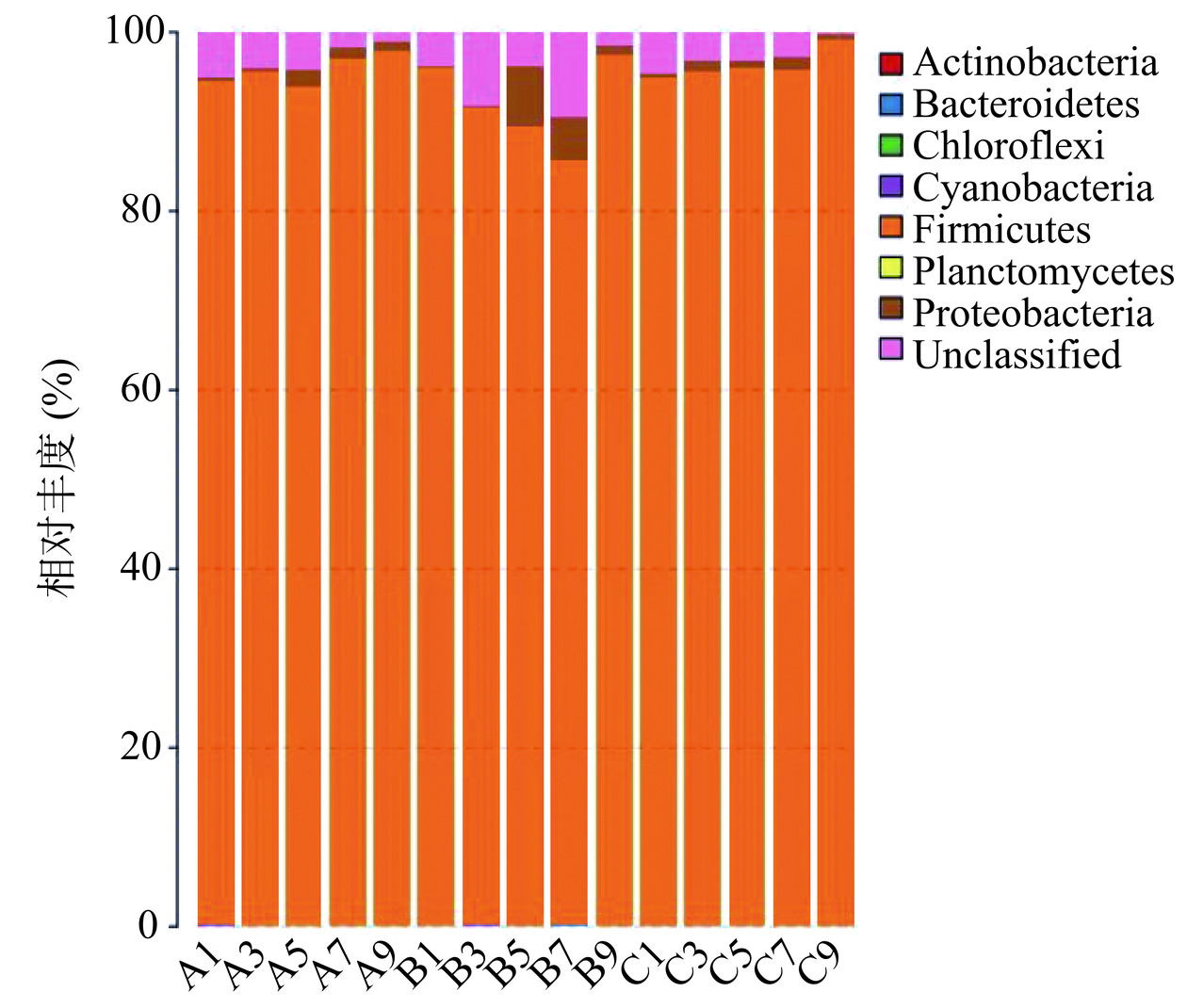
图3显示,参与山西老陈醋醋酸发酵阶段的细菌分属于7个门,其中厚壁菌门(Firmicutes)和变形菌门(Proteobacteria)是醋酸发酵阶段的优势菌,在醋醅样品中始终存在。厚壁菌门的细菌在数量上占有绝对的优势,#1,#3批次中,厚壁菌门的相对丰度均呈上升趋势,至发酵末期,其相对丰度甚至达到98%以上,在整个醋酸发酵阶段处于主导地位;第二批次(#2)中,其相对丰度虽然先降低后升高,但也一直维持在80%以上。随着醋酸发酵的进行,变形菌门的相对丰度呈缓慢增长趋势,至发酵第5 d(#1、#2)达到最大,随后其数量略有下降。除了厚壁菌门和变形菌门外,其他5个门(放线菌门Actinobacteria,拟杆菌门Bacteroidetes,绿弯菌门Chloroflexi,蓝藻细菌Cyanobacteria和浮霉菌门Planctomycetes)的相对丰度相对都很低,其中放线菌门和拟杆菌门在整个醋酸发酵发过程中一直存在。
2.4.2 醋酸发酵阶段细菌群落在属水平的结构分布以及动态变化
通过RDP classifer(v2.2)软件将OTU代表序列与数据库比对后,从图4可以看出,发酵前中期醋醅细菌的种类和丰度明显高于发酵末期的样本,这是因为酒化阶段的产物酒醪及拌醅加入的谷糠、麸皮和稻壳中都含有大量的微生物,且此时发酵底物充足,各种微生物快速的生长、繁殖、代谢;随着醋酸发酵的推进,微生物慢慢开始处于一个高温、高酸的胁迫环境,许多耐受能力差的微生物逐渐消亡,耐受性强的微生物保存下来,并通过增殖代谢,产生有机酸、酯类、醇类、氨基酸态氮等物质,完成醋酸发酵[25]。
乳杆菌属(Lactobacillus)和醋酸菌属(Acetobacter)的细菌在醋酸发酵细菌群落中占主导优势,发酵前5 d二者的丰度之和约占总数的90%左右,且乳酸菌的生物量远高于醋酸菌及其他细菌,与镇江香醋[25]及天津独流老醋[26]的免培养结果一致,说明不同地区参与醋酸发酵的优势细菌菌群结构大体相似。乳酸菌代谢产生的乳酸是食醋的第二大有机酸,可以缓解醋酸带来的刺激性酸味,改善食醋的口感。Jiang等[27]及余永建[28]的研究表明,食醋酿造中添加乳酸菌可以提高乳酸含量,改善食醋风味。在醋酸发酵阶段乳酸菌为丰度最高的细菌,在发酵前期,乳杆菌属的丰度为57.4%~66.4%左右,魏斯氏菌属(Weissella)的丰度为0.2%~1.8%左右。乳酸菌的生物量远超过其他细菌,是醋酸发酵阶段的优势菌群,其代谢物对食醋的风味物质具有重要的贡献[29]。在发酵前中期(第1~5 d),乳酸菌的丰度最高,可能有些乳酸菌来自酒精发酵的产物酒醪中。
三个发酵批次的前中期样本中,乳杆菌属、醋酸菌属及魏斯氏菌属(Weissella)为优势菌群,在第7 d和第9 d的醋醅样本中,三个属的微生物数量均明显减少。乳杆菌属的变化最为明显,发酵第7 d和第9 d,乳杆菌属的丰度大幅下降,分别降低21.4%~32.3%和4.1%~5.2%。发酵前期的醋醅样品中并未检测到假单胞菌属(Pseudomonas),但是随着醋酸发酵的进行,其在发酵第5 d的样本中出现,并在第7 d达到峰值,丰度1.8%,在第9 d样本中丰度又降为0.9%。乳酸含量随醋酸发酵的进行逐渐升高,假单胞菌是乳酸的主要利用者[30],发酵第5 d的醋醅中乳酸的含量为1.08 g/100 mL,推测乳酸只有积累到一定含量,假单胞菌才能开始增殖,随着醋醅总酸含量的升高,假单胞菌属因耐受不了高酸环境(总酸5.97 g/100 g)生物量逐渐减少。王丽娟[31]发现A. pasteurianus能利用乳酸代谢生成乙偶姻,其推测醋酸发酵过程中富含乳酸的酒醪可能是A. pasteurianus代谢生成乙偶姻的主要底物,而乳酸菌的存在也能为醋酸菌的代谢提供乳酸,进一步积累乙偶姻。醋酸发酵过程中还检测到有芽孢杆菌(Bacillus)的存在,有文献表明[32−33]芽孢杆菌可以产生川芎嗪以及其合成前体物质四甲基吡嗪,多种菌彼此互存、共同发酵,赋予食醋丰富的营养及一定的保健功效。
发酵过程中总酸的不断上升主要归功于醋酸的快速积累,而醋酸菌是乙酸的主要生产者[34]。山西老陈醋醋酸发酵阶段的醋酸菌均为醋杆菌属。随着发酵的进行,醋酸菌属的丰度从前期的29.6%降低到15.2%,在发酵结束时丰度降至5%左右。醋杆菌是食醋工业生产采用的主要醋酸菌,其主要功能是氧化糖和乙醇生成醋酸。本研究发现,醋酸菌的丰度远低于乳酸菌,但仍可以将大量的乙醇转化成醋酸,说明醋酸菌具有较高的氧化效率。魏斯氏菌属(Weissella)和链球菌属(Streptococcus)在整个醋酸发酵过程中都存在,其相对丰度在0.5%~2%,这两类菌虽然占比较小,但对风味物质的产生有一定的贡献作用。低丰度的微生物种类在不同地区的食醋酿造过程中不尽相同,乳球菌属(Lactococcus)和芽孢杆菌属(Bacillus)参与山西老陈醋和镇江香醋的发酵,而念珠藻属(Nostoc)、类诺卡氏菌属(Nocardioides)等是天津独流老醋特有的低丰度物种[26]。不同地区酿造食醋的风味各异固然与酿醋原料、加工工艺有关,但参与其中的微生物是各种物质的主要贡献者,微生物种类不同,代谢产物也大不一样。
2.5 物种Heatmap分析
Heatmap是一种根据物种种类或样品丰度的相似性进行的聚类分析[35],通过颜色梯度来展示矩阵中不同数值的大小,可以直观地展示不同醋醅样品间群落结构的相似性和差异性。图5是在属的分类等级进行的聚类分析,其中横向聚类表示同一物种在不同样本中的丰度相似情况,纵向聚类表示不同物种在所有样品间表达的相似情况,将所有样品中丰度均低于0.1%的物种合并成Others。图5显示,#1和#2的第1、3、5 d醋醅及#3第1、3 d醋醅样本的微生物物种组成及丰度较相似;而#1和#2的第7、9 d醋醅及#3第5、7、9 d醋醅样本的微生物物种组成及丰度较相似。颜色梯度表示丰度大小,红色越浅表示相对丰度越低,而绿色越浅则表示其相对丰度越高。因此,乳杆菌属(Lactobacillus)与醋酸菌属(Acetobacter)的细菌是醋酸发酵阶段相对丰度最高的微生物。
3. 结论
通过高通量测序分析,由于固态基质的复杂性和不均一性,随着醋酸发酵的进行,细菌的菌群丰富度和多样性也在不断变化。纵向比较,跟踪采样发现,同一批次不同发酵时间的醋醅样本,随着发酵的进行,其细菌群落虽处于不断的演替和变化中,但整个发酵阶段始终存在的物种占总物种的50%左右,促使发酵的有序进行;横向比较,三个批次相同发酵时间的醋醅样品中,共有OTU数目远大于各样品特有OTU的个数(共有OTU数目约占比总数的70%),说明不同批次间其细菌菌群结构具有很大的相似性,从而为不同批次产品质量的稳定性提供保障。
在属水平,乳杆菌属和醋酸菌属占主导优势,在发酵前5 d二者的相对丰度之和高达90%左右。除此以外,还有一些低丰度细菌包括魏斯氏菌属、链球菌属、芽孢杆菌属、假单胞菌属等适应醋酸发酵环境的微生物,也参与山西老陈醋的醋酸发酵过程中。这些微生物赋予了山西老陈醋独特的风味。
-
图 1 各批次醋酸发酵阶段OTU的Venn图
注:图中a、b、c代表三个发酵批次,d为三个批次的混合物。同样颜色代表不同发酵批次相同发酵时间的醋醅(如:A1、 B1、 C1)。A1、A3、…、A9表示同一发酵批次1、3、…、9 d的醋醅样本;AAF是将三个批次相同发酵时间的醋醅混合在一起;图2同。
Figure 1. Venn diagram of OTU in each batch of acetic acid fermentation stage
表 1 山西老陈醋不同批次醋醅取样时间及其编号
Table 1 Sampling times and batch numbers of Pei samples during acetic acid fermentation (AAF) of Shanxi aged vinegar
批次 取样时间 Day 1 Day 3 Day 5 Day 7 Day 9 混合 #1 A1 A3 A5 A7 A9 A #2 B1 B3 B5 B7 B9 B #3 C1 C3 C5 C7 C9 C 混合 AAF1 AAF3 AAF5 AAF7 AAF9 D 表 2 各样本中细菌群落测序情况以及在97%相似度下OTU划分情况
Table 2 Sequencing of bacterial communities and OTU division under 97% similarity in each Pei sample
样品名称 净序列 平均长度(bp) 有效序列(%) 序列数量 OTU数量(个) A1 88805 252 99.1 81013 78 A3 87778 252 99.27 82005 83 A5 88289 252 99.26 81198 95 A7 89346 252 99.11 84549 89 A9 87294 252 98.9 85046 71 B1 87886 252 98.99 81733 81 B3 88624 252 98.87 83436 80 B5 87933 252 99.12 82185 94 B7 88474 252 98.89 81332 95 B9 87894 252 98.63 85801 72 C1 87898 252 98.95 81356 86 C3 88418 252 98.96 80735 87 C5 88911 252 99.3 81611 98 C7 89143 252 99.22 83491 94 C9 87407 252 99.2 85763 66 表 3 醋醅样本中细菌群落的Alpha多样性
Table 3 Alpha diversity of bacterial community in Pei samples
样品名称 Sobs Chao指数 Ace指数 Shannon指数 Simpson指数 Coverage A1 86 77.60 76.88 2.14 0.18 99% A3 73 80.00 76.21 2.06 0.20 99% A5 76 86.00 86.18 2.26 0.16 99% A7 72 78.10 79.58 1.49 0.40 99% A9 69 92.00 93.70 0.64 0.79 99% B1 80 79.43 79.81 1.86 0.26 99% B3 75 103.25 101.24 2.14 0.18 99% B5 77 86.20 103.37 2.03 0.24 99% B7 73 80.17 80.72 2.20 0.17 99% B9 68 82.09 83.08 0.62 0.79 99% C1 85 96.00 87.23 1.89 0.24 99% C3 80 83.00 81.58 2.21 0.17 99% C5 72 100.17 97.76 2.26 0.15 99% C7 72 93.20 87.57 1.66 0.36 99% C9 61 69.67 71.82 0.47 0.84 99% -
[1] TING X, BO Z, WEN H D, et al. Hepatoprotective efficacy of Shanxi aged vinegar extract against oxidative damage in vitro and in vivo[J]. Journal of Functional Foods,2019,60(7):103448.
[2] TING X, JIN Z, JIA H Y, et al. Shanxi aged vinegar prevents alcoholic liver injury by inhibiting CYP2E1 and NADPH oxidase activities[J]. Journal of Functional Foods,2018,47(6):575−584.
[3] CHEN T, GUI Q, SHI J J, et al. Analysis of variation of main components during aging process of Shanxi aged vinegar[J]. Acetic Acid Bacteria,2013,2(1s):6. doi: 10.4081/aab.2013.s1.e6
[4] 邢晓莹. 基于山西老陈醋优良菌株在山楂醋酿造中的应用[D]. 晋中:山西农业大学, 2019. [XING X Y. Fermentation of hawthorn vinegar using excellent strains isolated from Shanxi aged vinegar[D]. Jinzhong:Shanxi Agricultural University, 2019.] XING X Y. Fermentation of hawthorn vinegar using excellent strains isolated from Shanxi aged vinegar[D]. Jinzhong: Shanxi Agricultural University, 2019.
[5] NIE Z, ZHENG Y, DU H, et al. Dynamics and diversity of microbial community succession in traditional fermentation of Shanxi aged vinegar[J]. Food Microbiology,2015,47(6):62−68.
[6] BO B, KIM S A, HAN N S. Bacterial and fungal diversity in Laphet, traditional fermented tea leaves in Myanmar, analyzed by culturing, DNA amplicon-based sequencing, and PCR-DGGE methods[J]. International Journal of Food Microbiology,2020,320:108508. doi: 10.1016/j.ijfoodmicro.2020.108508
[7] SUBASINGHE R M, SAMARAJEEWA A D, SCROGGINS R, et al. Evaluation of denaturing gradient gel electrophoresis (DGGE) and next generation sequencing (NGS) in combination with enrichment culture techniques to identify bacteria in commercial microbial-based products[J]. Journal of Microbiological Methods,2019,161:118−130. doi: 10.1016/j.mimet.2019.04.017
[8] JAMPAPHAENG K, COCOLIN L, MANEERAT S. Bacterial population diversity in Sataw-Dong, a traditional fermented stink bean, during fermentation using the combination of culturedependent and culture independent methods through DGGE technique[J]. Songklanakarin Journal of Science and Technology (SJST),2019,41(2):285−291.
[9] WANG J, HAO S, REN Q. Uncultured microorganisms and their functions in the fermentation systems of traditional Chinese fermented foods[J]. Foods,2023,12(14):2691. doi: 10.3390/foods12142691
[10] YUAN L, FAN L, LIU S, et al. Bacterial community analysis of infant foods obtained from Chinese markets by combining culture-dependent and high-throughput sequence methods[J]. Food Research International,2022,162:112060. doi: 10.1016/j.foodres.2022.112060
[11] KAVITAKE D, SURYAVANSHI M V, KANDASAMY S, et al. Bacterial diversity of traditional fermented food, Idli by high thorough-put sequencing[J]. Journal of Food Science and Technology,2022,59(10):3918−3927. doi: 10.1007/s13197-022-05421-4
[12] LIANG H, CHEN H, JI C, et al. Dynamic and functional characteristics of predominant species in industrial paocai as revealed by combined DGGE and metagenomic sequencing[J]. Frontiers in Microbiology,2018,9:2416. doi: 10.3389/fmicb.2018.02416
[13] SHANGPLIANG H N J, TAMANG J P. Metagenomics and metagenome-assembled genomes mining of health benefits in Jalebi batter, a naturally fermented cereal-based food of India[J]. Food Research International,2023,172:113130. doi: 10.1016/j.foodres.2023.113130
[14] 范三红, 薛虎贵, 白宝清, 等. 山西陈醋发酵过程微生物群落动态分析及差异菌属筛选[J]. 食品工业科技,2022,43(24):171−179. [FAN S H, XUE H G, BAI B Q, et al. Dynamic analysis of microbial community and selection of different genera during fermentation of Shanxi vinegar[J]. Science and Technology of Food Industry,2022,43(24):171−179.] FAN S H, XUE H G, BAI B Q, et al. Dynamic analysis of microbial community and selection of different genera during fermentation of Shanxi vinegar[J]. Science and Technology of Food Industry, 2022, 43(24): 171−179.
[15] 杨玲, 刘春, 彭佳伟, 等. 山西老陈醋和怀仁醋酒精发酵阶段细菌菌群多样性分析[J]. 中国酿造,2023,42(2):63−69. [YANG L, LIU C, PENG J W, et al. Bacterial diversity analysis of Shanxi aged vinegar and Huairen vinegar during alcohol fermentation[J]. China Brewing,2023,42(2):63−69.] YANG L, LIU C, PENG J W, et al. Bacterial diversity analysis of Shanxi aged vinegar and Huairen vinegar during alcohol fermentation[J]. China Brewing, 2023, 42(2): 63−69.
[16] ZHU Y P, ZHANG F F, ZHANG C N, et al. Dynamic microbial succession of Shanxi aged vinegar and its correlation with flavor metabolites during different stages of acetic acid fermentation[J]. Scientific Reports,2018,8(1):1−10.
[17] NIE Z Q, ZHENG Y, XIE S K, et al. Unraveling the correlation between microbiota succession and metabolite changes in traditional Shanxi aged vinegar[J]. Scientific Reports,2017,7(1):9240. doi: 10.1038/s41598-017-09850-6
[18] LI S, LI P, LIU X, et al. Bacterial dynamics and metabolite changes in solid-state acetic acid fermentation of Shanxi aged vinegar[J]. Appl Microbiol Biotechnol,2016,100(10):4395−4411. doi: 10.1007/s00253-016-7284-3
[19] 孙蒙, 刘璐, 谭心, 等. 高通量测序解析契达奶酪在加工过程中菌群结构多样性变化[J]. 食品工业科技,2024,45(6):161−168. [SUN M, LIU L, TAN X, et al. High-throughput sequencing to analyze changes in the structural diversity of the flora of Cheddar cheese during processing[J]. Science and Technology of Food Industry,2024,45(6):161−168.] SUN M, LIU L, TAN X, et al. High-throughput sequencing to analyze changes in the structural diversity of the flora of Cheddar cheese during processing[J]. Science and Technology of Food Industry, 2024, 45(6): 161−168.
[20] MAGOC T, SALZBERG S L. Flash:Fast length adjustment of short reads to improve genome assemblies[J]. Bioinformatics,2011,27(21):2957−2963. doi: 10.1093/bioinformatics/btr507
[21] EDGAR R C. Uparse:Highly accurate OTU sequences from microbial amplicon reads[J]. Nature Methods,2013,10(10):996−998. doi: 10.1038/nmeth.2604
[22] 赵烜影, 苑秀娟, 郭鸰, 等. 传统自然发酵黏豆包面团微生物菌群结构分析[J]. 食品科学,2018,39(18):67−72. [ZHAO X Y, YUAN X J, GUO L, et al. Microbial community composition of traditional spontaneously fermented sticky bean bun dough[J]. Food Science,2018,39(18):67−72.] doi: 10.7506/spkx1002-6630-201818011 ZHAO X Y, YUAN X J, GUO L, et al. Microbial community composition of traditional spontaneously fermented sticky bean bun dough[J]. Food Science, 2018, 39(18): 67−72. doi: 10.7506/spkx1002-6630-201818011
[23] 叶长文, 李璐, 贺琛, 等. 基于高通量测序的雪茄烟微生物群落结构和多样性分析[J]. 烟草科技,2021,54(8):1−9. [YE C W, LI L, HE C, et al. Structure and diversity analysis of microbial communities in cigar products by high-throughput sequencing technology[J]. Tobacco Science & Technology,2021,54(8):1−9.] YE C W, LI L, HE C, et al. Structure and diversity analysis of microbial communities in cigar products by high-throughput sequencing technology[J]. Tobacco Science & Technology, 2021, 54(8): 1−9.
[24] 倪霞, 龚林, 陈华, 等. 基于高通量测序的昭通核心烟区植烟土壤真菌多样性分析[J]. 中国农学通报,2024,40(2):61−70. [NI X, GONG L, CHEN H, et al. Analysis of soil fungal diversity in Zhaotong core tobacco-growing area based on high-throughput sequencing[J]. Chinese Agricultural Science Bulletin,2024,40(2):61−70.] doi: 10.11924/j.issn.1000-6850.casb2023-0087 NI X, GONG L, CHEN H, et al. Analysis of soil fungal diversity in Zhaotong core tobacco-growing area based on high-throughput sequencing[J]. Chinese Agricultural Science Bulletin, 2024, 40(2): 61−70. doi: 10.11924/j.issn.1000-6850.casb2023-0087
[25] 王宗敏. 镇江香醋醋酸发酵阶段菌群结构变化与风味物质组成之间的相关性研究[D]. 无锡:江南大学, 2016. [WANG Z M. Correlation between structural shifts of microbiota and compositions of flavors during the stage of acetic acid fermentation of Zhenjiang aromatic vinegar[D]. Wuxi:Jiangnan University, 2016.] WANG Z M. Correlation between structural shifts of microbiota and compositions of flavors during the stage of acetic acid fermentation of Zhenjiang aromatic vinegar[D]. Wuxi: Jiangnan University, 2016.
[26] 聂志强, 韩玥, 郑宇, 等. 宏基因组学技术分析传统食醋发酵过程微生物多样性[J]. 食品科学,2013,34(15):198−203. [NIE Z Q, HAN Y, ZHENG Y, et al. Metagenomic analysis of microbial diversity in the traditional vinegar fermentation process[J]. Food Science,2013,34(15):198−203.] doi: 10.7506/spkx1002-6630-201315041 NIE Z Q, HAN Y, ZHENG Y, et al. Metagenomic analysis of microbial diversity in the traditional vinegar fermentation process[J]. Food Science, 2013, 34(15): 198−203. doi: 10.7506/spkx1002-6630-201315041
[27] JIANG Y, GUO J, LI Y, et al. Optimisation of lactic acid fermentation for improved vinegar flavour during rosy vinegar brewing[J]. Journal of the Science of Food and Agriculture,2010,90(8):1334−1339. doi: 10.1002/jsfa.3986
[28] 余永建. 镇江香醋有机酸组成及乳酸合成的生物强化[D]. 无锡:江南大学, 2014. [YU Y J. Organic acids analysis and enhanced lactic acid synthesis of Zhenjiang aromatic vinegar[D]. Wuxi:Jiangnan University, 2014.] YU Y J. Organic acids analysis and enhanced lactic acid synthesis of Zhenjiang aromatic vinegar[D]. Wuxi: Jiangnan University, 2014.
[29] RONG K, MIN L, JUNDE X, et al. Exploring of seasonal dynamics of microbial community in multispecies fermentation of Shanxi mature vinegar[J]. Journal of Bioscience and Bioengineering,2022,133(4):375−381. doi: 10.1016/j.jbiosc.2022.01.003
[30] 吴林寰. 整合型元基因组序列分析平台构建及其在酿醋微生物群落功能研究中的应用[D]. 无锡:江南大学, 2016. [WU L H. Construction of integrated metagenomics analysis platform and its application on functional study of vinegar microbiota[D]. Wuxi:Jiangnan University, 2016.] WU L H. Construction of integrated metagenomics analysis platform and its application on functional study of vinegar microbiota[D]. Wuxi: Jiangnan University, 2016.
[31] 王丽娟. 产乙偶姻功能微生物群落的分离及其在食醋酿造中的应用[D]. 无锡:江南大学, 2018. [WANG L J. Isolation of acetoin-producing microbial community in vinegar Pei and its application in vinegar brewing process[D]. Wuxi:Jiangnan University, 2018.] WANG L J. Isolation of acetoin-producing microbial community in vinegar Pei and its application in vinegar brewing process[D]. Wuxi: Jiangnan University, 2018.
[32] KOSUGE T, ADACHI T, KAMIYA H. Isolation of tetramethylpyrazine from culture of Bacillus natto, and biosynthetic pathways of tetramethylpyrazine[J]. Nature,1962,195(4846):1103.
[33] 赵德义, 汤丹丹, 曹建全, 等. 产四甲基吡嗪微生物菌株的选育[J]. 中国酿造,2015,34(3):102−106. [ZHAO D Y, TANG D D, CAO J Q, et al. Screening of tetramethylpyrazine-producing strain[J]. China Brewing,2015,34(3):102−106.] doi: 10.11882/j.issn.0254-5071.2015.03.024 ZHAO D Y, TANG D D, CAO J Q, et al. Screening of tetramethylpyrazine-producing strain[J]. China Brewing, 2015, 34(3): 102−106. doi: 10.11882/j.issn.0254-5071.2015.03.024
[34] 魏世东, 王素英, 娄婷婷, 等. 食醋中挥发性风味物质及其检测方法研究进展[J]. 食品研究与开发,2022,43(24):215−224. [WEI S D, WANG S Y, LOU T T, et al. Progress of research on volatile flavor substances in vinegar and their detection methods[J]. Food Research and Development,2022,43(24):215−224.] doi: 10.12161/j.issn.1005-6521.2022.24.029 WEI S D, WANG S Y, LOU T T, et al. Progress of research on volatile flavor substances in vinegar and their detection methods[J]. Food Research and Development, 2022, 43(24): 215−224. doi: 10.12161/j.issn.1005-6521.2022.24.029
[35] 张波, 刘珂轶, 张安龙, 等. 不同营养环境下棕鞭藻共栖细菌的群落结构比较[J]. 环境科学学报,2020,40(8):2812−2818. [ZHANG B, LIU K Y, ZHANG A L, et al. Comparison of community structure of symbiotic bacteria of Ochromonas in different nutritional environments[J]. Acta Scientiae Circumstantiae,2020,40(8):2812−2818.] ZHANG B, LIU K Y, ZHANG A L, et al. Comparison of community structure of symbiotic bacteria of Ochromonas in different nutritional environments[J]. Acta Scientiae Circumstantiae, 2020, 40(8): 2812−2818.





 下载:
下载:


 下载:
下载:
